Мейзу и 10: Ваш браузер устарел — Москва
Meizu U10 — характеристики
Компания Мейзу не славилась огромным изобилием различных моделей. Но вслед за расширением модельного ряда другими производителями, она также решила выпустить несколько новых моделей. В частности, линейка представлена пополнением Meizu U10 и Meizu U20. Речь пойдет о U10, который отличается меньшей диагональю, соответственно и доступной стоимостью.
Дизайн
Речь идет не о флагмане, но компания постаралась, чтобы дизайн был флагманским. Фишкой данного гаджета стал притягательный дизайн, в котором сочетаются металл, стекло и глянец. В целом, с дизайном у компании итак никогда не было особых проблем: смартфоны от Мейзу и раньше выглядели неплохо, теперь же они перешли на другой уровень. Возможно, смартфон выглядит притягательно за счет того, что 2.5-д стекло покрывает не только лицевую панель, но и заднюю поверхность. Но в отличие от многих конкурентов, стекло устойчиво к царапина и практически не собирает отпечатки за счет применения олеофобного покрытия.
Завершая описание дизайнерских особенностей, нельзя не отметить довольно нетипичное расположение 3.5-мм разъема, который пользователи обнаружат не сверху, а на нижнем торце, где располагается микроюсб-разъем.
Начинка
Что ж, оформление у смартфона хорошее, а как быть с внутренней начинкой? Здесь все немного скромнее: устройство оснащается процессором медиатековского производства начального уровня – МТ6750. Конечно, для повседневных задач этого вполне хватит, тем более что в U10 установлено 2 или 3гб ОЗУ, в зависимости от выбора. Относительно ресурсоемкие 3д-приложения, конечно же, запускаются, пусть и не на максимальных настройках. Что до камеры, здесь использован тринадцатимегапиксельный модуль, аналогичный таковым в остальных бюджетниках компании.
Конечно, для повседневных задач этого вполне хватит, тем более что в U10 установлено 2 или 3гб ОЗУ, в зависимости от выбора. Относительно ресурсоемкие 3д-приложения, конечно же, запускаются, пусть и не на максимальных настройках. Что до камеры, здесь использован тринадцатимегапиксельный модуль, аналогичный таковым в остальных бюджетниках компании.
Meizu U10 — описание телефона
Основные характеристики телефона
Meizu U10
| Класс | смартфон, мультимедийный |
| Год выпуска | 2016, август |
| Стандарт | GSM 850/900/1800/1900-SIM 1 и SIM 2, HSDPA, LTE |
| Аккумулятор | Несъемный, Li-Ion 2760 мАч |
| Дисплей | 5,0″ IPS LCD, 720 x 1280 точек |
| Вес | 139 грамм |
| Размеры | 141,9×69,6×7,9 мм |
Подробные характеристики U10
Дополнительные характеристики Meizu U10
- Цвета: Черный, белый, золотой
- Встроенная камера: 13 Mп камера, автофокус с определением фазы, двойная светодиодная вспышка (двухтональная), 1080р-видео; фронтальная камера 5 Мп
- Другое: ОС Google Android 6.
 0 (Marshmallow)
Два четырехъядерных процессора с частотой 1,5 и 1 ГГц
0 (Marshmallow)
Два четырехъядерных процессора с частотой 1,5 и 1 ГГц
- Память
- 16/ 32 Гб встроенной памяти
- 2/ 3 Гб оперативной памяти
- Звонок, набор номера
- Виброзвонок
- Полифония
- Коммуникационные возможности
- GPRS/EDGE
- HSDPA/HSUPA
- LTE
- Wi-Fi 802.11 b/g/n, hotspot
- Bluetooth v4.0
- Разъем 3,5мм
- A-GPS, GLONASS
- Органайзер и дополнительные функции
- Аудио/видео плееры
- Акселерометр
- Датчик отпечатков пальцев
- Гироскоп
- Датчик положения
- Датчик освещенности
- Датчик приближения
- Компас

Meizu U10 — обзор, характеристики, отзывы, цены
Обзор | Характеристики | Цены | Отзывы
Обзор Meizu U10
Meizu U10 — «стеклянный» смартфон в компактном корпусе. Аппарат заполучил привлекательный дизайн, который является главной «фишкой» данного устройства. Сам гаджет был анонсирован в августе 2016 года.
Внешний вид и эргономика
Корпус Мейзу У10 реализован из металла и стекла. Так, с обеих сторон смартфона располагаются стеклянные 2,5D-панели, соединенные алюминиевой рамкой по периметру. Особую элегантность гаджету придает скругленный кант. Под экраном находится навигационная кнопка mTouch, совмещенная со сканером отпечатков. На верхнем торце нет абсолютно ничего. А вот нижняя грань получила аудио разъем, внешний динамик, микрофон и USB-порт. На левой стороне находится универсальный слот для карты памяти и двух сим-карт.
Что касается надежности корпуса, то закаленное стекло является достаточно прочным. Отсутствуют различные скрипы, что говорит об отличном качестве сборки. Из-за скользкого материала смартфон может съехать с неровной поверхности — это нужно учитывать во время использования. Модель снабжается аккумулятором с емкостью 2760 мАч. Доступные цвета: белый, черный, розовый и золотой. Габариты: высота — 141,9 мм, толщина — 7,9 мм, ширина — 69,6 мм, вес — 139 г.
Отсутствуют различные скрипы, что говорит об отличном качестве сборки. Из-за скользкого материала смартфон может съехать с неровной поверхности — это нужно учитывать во время использования. Модель снабжается аккумулятором с емкостью 2760 мАч. Доступные цвета: белый, черный, розовый и золотой. Габариты: высота — 141,9 мм, толщина — 7,9 мм, ширина — 69,6 мм, вес — 139 г.
Дисплей
U10 оснащается 5-дюймовым экраном с технологией полного ламинирования. При этом качественная матрица IPS имеет разрешение 1280 на 720 точек, что представляется классическим решением для подобной диагонали. Здесь нет воздушной прослойки, поэтому картинка выглядит очень натурально. Дисплей поддерживает десять одновременных касаний, а также выделяется максимальными углами обзора. Высокие показатели контрастности (1000:1) дополняются довольно неплохим уровнем максимальной яркости. Если же цветовая передача не устраивает, то ее можно отрегулировать самостоятельно.
Железо и производительность
В смартфоне У10 используется процессор MediaTek MT6750 с восемью ядрами и предельной тактовой частотой 1500 МГц. Роль графического ускорителя выполняет GPU Mali-T860. Тут есть 32 Гб встроенной памяти с возможностью увеличения объема до 128 Гб, а также 3 Гб ОЗУ LPDDR3. Существует и более простая версия с 2 Гб оперативной памяти и 16 Гб внутреннего хранилища.
Роль графического ускорителя выполняет GPU Mali-T860. Тут есть 32 Гб встроенной памяти с возможностью увеличения объема до 128 Гб, а также 3 Гб ОЗУ LPDDR3. Существует и более простая версия с 2 Гб оперативной памяти и 16 Гб внутреннего хранилища.
Гаджет поставляется вместе с операционной системой Android 6.0 и оболочкой Flyme. Так как смартфон относится к бюджетному классу, то 40000 баллов в тесте AnTuTu — вполне хороший результат. Под нагрузкой корпус нагревается равномерно, причем не очень сильно. Во многие простые игры можно поиграть без каких-либо проблем. «Тяжелые» проекты также запускаются, но придется понизить качество графики.
Связь и звук
Meizu U10 не может похвастаться идеальным звучанием, но и одновременно не разочаровывает. Умеренная громкость внешнего динамика дополняется хорошей проработкой многих частот. Разговорный динамик здесь весьма качественный, собеседника отчетливо слышно.
Камера
В Мейзу У10 располагается 13-мегапиксельная камера со светосилой 2,2 и светодиодной вспышкой двух тонов. Здесь есть и фазовый автофокус, отличающийся быстротой работы. Фронтальная камера является 5-мегапиксельной, имеет светосилу 2,0 и технологию Beauty features. Присутствует не только автоматический, но и ручной режим съемки.
Здесь есть и фазовый автофокус, отличающийся быстротой работы. Фронтальная камера является 5-мегапиксельной, имеет светосилу 2,0 и технологию Beauty features. Присутствует не только автоматический, но и ручной режим съемки.
Выводы
Элегантный и стильный смартфон U10 выглядит по-настоящему эффектно. Именно во внешнем виде скрывается все его очарование. При этом начинка аппарата вполне соответствует современному бюджетному классу. Комплектация: телефон, документация, скрепка, зарядное устройство, кабель USB-microUSB.
Плюсы:
- Замечательный дизайн из металла и стекла.
- Очень качественные материалы.
- Точный и быстрый сканер отпечатков.
- Эффективное олеофобное покрытие с двух сторон.
- Достаточно скромная цена за имиджевый гаджет.
Минусы:
- Не все частоты LTE поддерживаются.
- Корпус может показаться немного скользким.
Технические характеристики Meizu U10
| Общие характеристики | |
|---|---|
| Модель | Meizu U10 |
| Дата анонса и начала продаж | август 2016 / август 2016 |
| Размеры (ДxШxВ) | 141. 9 x 69.6 x 7.9 мм. 9 x 69.6 x 7.9 мм. |
| Вес | 139 г. |
| Доступные цвета | черный, белый, розовый и золото шампанского |
| Операционная система | Android 6.0 + Flyme |
| Связь | |
| Количество и тип SIM-карт | две, Nano-SIM |
| Стандарт связи в 2G сетях | GSM 850 / 900 / 1800 / 1900 — SIM 1 & SIM 2 |
| Стандарт связи в 3G сетях | HSDPA 850 / 900 / 1900 / 2100 |
| Стандарт связи в 4G сетях | LTE |
| Совместимость с операторами связи | МТС, Билайн, Мегафон, Tele2, Yota |
| Передача данных | |
| Wi-Fi | Wi-Fi 802.11 b/g/n, hotspot |
| Bluetooth | 4.0, A2DP, LE |
| GPS | есть, A-GPS, GLONASS |
| NFC | нет |
| Инфракрасный порт | нет |
| Платформа | |
| Процессор | восьмиядерный Mediatek MT6750 Octa-core (4×1.  5 GHz Cortex-A53 & 4×1.0 GHz Cortex-A53) 5 GHz Cortex-A53 & 4×1.0 GHz Cortex-A53) |
| Графический процессор | Mali-T860MP2 |
| Внутренняя память | 16 GB / 32 GB |
| Оперативная память | 2 GB / 3 GB |
| Порты и разъемы | |
| USB | microUSB 2.0 |
| 3.5mm jack | есть |
| Слот для карт памяти | microSD, до 128 GB (совмещенный со слотом SIM 2) |
| Дисплей | |
| Тип дисплея | IPS LCD емкостный, 16M цветов |
| Размер экрана | 5 дюймов (~69.8% лицевой поверхности устройства) |
| Защита дисплея | закаленное стекло |
| Камера | |
| Основная камера | 13 MP, f/2.2, фазовый автофокус, dual-LED (dual tone) вспышка |
| Функционал основной камеры | Geo-tagging, touch focus, face detection, HDR, panorama |
| Запись видео | 1080p@30fps |
| Фронтальная камера | 5 MP, f/2. 0 0 |
| Датчики и сенсоры | |
| Освещенности | есть |
| Приближения | есть |
| Гироскоп | есть |
| Компас | есть |
| Холла | нет |
| Акселерометр | есть |
| Барометр | нет |
| Сканер отпечатка пальца | есть |
| Батарея | |
| Тип и емкость аккумулятора | Li-Ion 2760 mAh |
| Крепление аккумулятора | несъемный |
| Комплектация | |
| Стандартный комплект | U10: 1 USB-кабель: 1 Скрепка для извлечения sim-лотка: 1 Руководство пользователя: 1 Гарантийный талон: 1 Зарядное устройство: 1 |
Цены
Видеообзоры
youtube.com/embed/-XU5S700kXc?feature=oembed» frameborder=»0″ allow=»autoplay; encrypted-media» allowfullscreen=»»/>
сравнить — 3 Гб оперативной памяти, 8 мпикс фронтальная камера Платформа Android 8.1 1500 Мгц 4-ядерный процессор + граф.процессор HD разрешение экрана (1440×720) | сравнить — Отделка кожей, 10000 мА*ч Li-Pol аккумулятор 5 мпикс фронтальная камера, платформа Android 7. 0 01500 Мгц 4-ядерный процессор + граф.процессор Встроенный сканнер отпечатка пальца Нецарапающееся стекло, 2 Гб оперативной памяти | сравнить — 3 Гб оперативной памяти, 5 мпикс фронтальная камера 1400 Мгц 4-ядерный процессор + граф. Встроенный сканнер отпечатка пальца HD разрешение экрана (1280×720) 13 мегапиксельная камера, стандарт связи LTE (4G) | ||||||||||||
сравнить от 7 700 р. 8 мпикс фронтальная камера 1500 Мгц 8-ядерный процессор + граф. Встроенный сканнер отпечатка пальца 3070 мА*ч Li-Ion аккумулятор | сравнить — 5 мпикс фронтальная камера, платформа Android 7. 1400 Мгц 4-ядерный процессор + граф.процессор Встроенный сканнер отпечатка пальца Нецарапающееся стекло, 2 Гб оперативной памяти 3080 мА*ч Li-Ion аккумулятор | сравнить — 4 Гб оперативной памяти 2500 Мгц 8-ядерный процессор + граф. 5 мпикс фронтальная камера, платформа Android 7.0 Встроенный сканнер отпечатка пальца Нецарапающееся стекло, 64 Гб встроенной памяти | ||||||||||||
сравнить — 6 Гб оперативной памяти 2500 Мгц 8-ядерный процессор + граф. 5 мпикс фронтальная камера, платформа Android 7.0 128 Гб встроенной памяти Встроенный сканнер отпечатка пальца | сравнить от 9 990 р. Водонепроницаемый и пыленепроницаемый корпус 8 мпикс фронтальная камера, платформа Android 6. 1600 Мгц 8-ядерный процессор + граф.процессор Встроенный сканнер отпечатка пальца Нецарапающееся стекло, 2 Гб оперативной памяти | сравнить от 9 390 р. 3 Гб оперативной памяти, 5 мпикс фронтальная камера Платформа Android 6. 1400 Мгц 8-ядерный процессор + граф.процессор Встроенный сканнер отпечатка пальца 3020 мА*ч Li-Ion аккумулятор | ||||||||||||
сравнить от 5 990 р. 5 мпикс фронтальная камера, платформа Android 6. 1500 Мгц 8-ядерный процессор + граф.процессор Встроенный сканнер отпечатка пальца Нецарапающееся стекло, 2 Гб оперативной памяти 3070 мА*ч Li-Ion аккумулятор | сравнить — 3 Гб оперативной памяти, 5 мпикс фронтальная камера Платформа Android 6. 1500 Мгц 8-ядерный процессор + граф.процессор 4100 мА*ч Li-Ion аккумулятор Встроенный сканнер отпечатка пальца | сравнить от 9 990 р. 5 мпикс фронтальная камера, платформа Android 6. 1400 Мгц 4-ядерный процессор + граф.процессор Встроенный сканнер отпечатка пальца 2 Гб оперативной памяти HD разрешение экрана (1280×720) | ||||||||||||
сравнить от 11 990 р. 3 Гб оперативной памяти, 5 мпикс фронтальная камера Платформа Android 6. 1500 Мгц 8-ядерный процессор + граф.процессор Встроенный сканнер отпечатка пальца Нецарапающееся стекло, HD разрешение экрана (1280×720) | сравнить от 13 990 р. 3 Гб оперативной памяти, 5 мпикс фронтальная камера Платформа Android 5. 1800 Мгц 8-ядерный процессор + граф.процессор Встроенный сканнер отпечатка пальца Нецарапающееся стекло, 3260 мА*ч Li-Ion аккумулятор | сравнить от 8 570 р. 5 мпикс фронтальная камера, платформа Android 6. 1800 Мгц 8-ядерный процессор + граф.процессор Встроенный сканнер отпечатка пальца Нецарапающееся стекло, 2 Гб оперативной памяти 3260 мА*ч Li-Ion аккумулятор | ||||||||||||
| |||||
| |||||
| |||||
| |||||
| |||||
| |||||
| |||||
| |||||
| |||||
| |||||
| |||||
| |||||
| |||||
| |||||
| |||||
| |||||
| |||||
| |||||
| |||||
| |||||
| |||||
| |||||
| |||||
Meizu U10 32GB Black технические характеристики смартфона Meizu U10 32GB Black
Общая информация
Производитель
Meizu
Общие характеристики
Стандарт
GSM 850/900/1800/1900, 3G, LTE (4G)
Платформа
Android
Операционная система
Android 6. 0
0
Тип корпуса
моноблок
Количество SIM-карт
2 сим карты
Тип сенсорного экрана
емкостный (multitouch)
Диагональ ?
5 дюймов
Размер изображения
1280×720
Автоматический поворот экрана
есть
Мультимедийные возможности
Фотокамера
13 млн пикс. , светодиодная вспышка
, светодиодная вспышка
Фронтальная камера
есть, 5 млн пикс.
Функции камеры
автофокус
Разъем для наушников
3. 5 мм
5 мм
Память и процессор
Поддержка карт памяти
microSD
Объем оперативной памяти (RAM)
3 Гб
Объем постоянной памяти (ROM)
32 Гб
Процессор ?
MediaTek MT6750
Количество ядер процессора
8
Интерфейсы ?
Bluetooth, Wi-Fi
Встроенный GPS-приемник
есть
Cистема A-GPS
есть
Доступ в интернет
3G, GPRS/EDGE, LTE (4G), Wi-Fi
Емкость аккумулятора
2760 мАч
Другие функции
Громкая связь (встроенный динамик)
есть
Управление
голосовой набор, голосовое управление
Режим полета
есть
Датчики
Освещенности, приближения, гироскоп, компас, считывание отпечатка пальца
Физические параметры (нетто)
Размеры (ШxВxГ)
69,6 х 141,9 х 7,9 мм
Вес (нетто)
139 г
Физические параметры (брутто)
Размеры (брутто, см)
15. 1×8.02×6.19
1×8.02×6.19
Вес (брутто, кг)
0.33
Обзор Meizu U10: доступный смартфон с флагманским дизайном
Под конец лета 2016 года компания Мейзу представила свою новую линейку смартфонов, в лице U10 и U20. Это недорогие устройства, с привычным для своей ценовой категории железом. Их главная фишка – это притягательный дизайн, с глянцевым корпусом из металла и стекла. В этом обзоре пойдет речь о Meizu U10, который обладает меньшим дисплеем (а значит и габаритами), и заодно стоит дешевле.
За конфигурацию с объемами памяти 2/16 ГБ просят от 148 USD, и от 167 USD за вариант с 3/32 ГБ. Указанные цены примерные, и актуальные на момент создания этого обзора. Остается выяснить, что представляет собой смартфон от Meizu, стоимостью менее 200 долларов.
Указанные цены примерные, и актуальные на момент создания этого обзора. Остается выяснить, что представляет собой смартфон от Meizu, стоимостью менее 200 долларов.
Характеристики Meizu U10
Технические характеристики почти полностью идентичны Meizu M3s.
Внешний вид, разъемы, кнопки и датчики
Meizu U10 получил полностью переосмысленный дизайн. Да, раньше смартфоны этого производителя тоже выглядели неплохо, но теперь это совершенно иной уровень. Лицевую и тыльную панель полностью покрывает скругленное 2.5D стекло.
Оно устойчиво к царапинам, и покрыто олеофобным слоем, благодаря которому пальцы легко скользят по его поверхности.
Рамки из металла получили прямоугольную форму, и на них появились рельефные скошенные края, подчеркивающие природу материала.
Доступны такие цвета корпуса: черный, белый, золотистый, и розовый. Независимо от расцветки, панель будет зеркальная и глянцевая. В черном цвете аппарат выглядит просто шикарно. Правда, ровно до тех пор, пока не прикасаться к нему. Даже после непродолжительного использования на корпус страшно смотреть из-за множества отпечатков и жирных пятен.
Правда, ровно до тех пор, пока не прикасаться к нему. Даже после непродолжительного использования на корпус страшно смотреть из-за множества отпечатков и жирных пятен.
Олеофобный слой попросту не справляется с ними. Радует, что на панели другого цвета (белого, например) такого безобразия почти не видно.
Расположение элементов вполне привычное, не считая разъема 3.5 мм. Лоток для установки СИМ-карт и накопителя MicroSD находится на левой боковой грани, на правой расположились кнопки питания и громкости. Верхнее ребро пустует, зато на нижнем поместился разъем MicroUSB с поддержкой OTG, стандартный аудиоджек, отверстия основного динамика и микрофон.
Привычных сенсорных кнопок для навигации ни на панели, ни на экране здесь нет. Вместо них под экраном установлена универсальная кнопка mTouch 2.1, сенсорная и механическая одновременно. Используется для навигации в интерфейсе ОС, имеет встроенный сканер отпечатков пальцев. Запоминает до 5 отпечатков, угол распознавания достигает 360 градусов. Датчик не настолько быстрый, как у флагманов, но скорость и точность срабатывания более, чем удовлетворительная. Габариты изделия: 141.9 x 69.6 x 7.9 мм, его масса – 139 г.
Датчик не настолько быстрый, как у флагманов, но скорость и точность срабатывания более, чем удовлетворительная. Габариты изделия: 141.9 x 69.6 x 7.9 мм, его масса – 139 г.
Процессор
Смартфон оснащается 8-ядерным процессором начального уровня MediaTek 6750, с архитектурой ARM big.LITTLE. Ядра Cortex-A53 разбиты на 2 кластера: 4 из них работают с частотой 1.5 ГГц, а оставшиеся 4 ограничены частотой 1 ГГц. Роль графического ускорителя выполняет чип Mali-T860. Результаты синтетического теста Антуту: от 31 до 40 тыс. баллов. В данном примере показатели зависят от версии прошивки, и от объема оперативки.
На практике смартфон уверенно справляется с тривиальными задачами. Нагрузить аппарат и заставить задумываться, конечно, можно. Но для устройства подобной ценовой категории это вполне нормально. Запускаются любые ресурсоемкие 3D игр, хоть и не всегда на максимальном уровне графики. При интенсивных нагрузках проблем с избыточным нагревом корпуса не наблюдается.
Память
Смартфон поставляется в двух конфигурациях: 2 либо 3 ГБ ОЗУ, и 16 либо 32 ГБ ПЗУ. Что касается оперативной памяти, то версия с 3 ГБ ОЗУ продемонстрирует чуть лучшую производительность в играх. А для многозадачности останется чуть больше свободной памяти. То есть, можно спокойно пользоваться версией на 2 ГБ, уровня ее быстродействия за глаза достаточно для не особо требовательных пользователей.
Что касается оперативной памяти, то версия с 3 ГБ ОЗУ продемонстрирует чуть лучшую производительность в играх. А для многозадачности останется чуть больше свободной памяти. То есть, можно спокойно пользоваться версией на 2 ГБ, уровня ее быстродействия за глаза достаточно для не особо требовательных пользователей.
Со встроенным накопителем дела обстоят следующим образом. Если нужно много свободного места, и планируется использование двух СИМ – придется брать конфигурацию с 32 ГБ. Если хочется немного сэкономить, и есть возможность обойтись без дополнительной карты SIM, очевидно, подойдет и вариант с 16 ГБ. В таком случае, пространство легко расширить до 128 ГБ с помощью внешнего MicroSD накопителя.
Автономная работа
В начале обзора упоминалось о поразительном сходстве между начинкой U10 и M3s. Так вот, аккумулятор – это исключение: с 3020 мАч емкость снизилась до 2760 мАч. Единственным логическим объяснением подобного даунгрейда можно считать попытку производителя повлиять на габариты. Как результат – толщина корпуса U10 составляет 7.9 мм против 8.3 мм у M3s.
Как результат – толщина корпуса U10 составляет 7.9 мм против 8.3 мм у M3s.
При среднестатистической интенсивности использования батарея выдерживает 1 день. Чтобы при таком же режиме эксплуатации добиться работы на протяжении суток, придется научиться экономить. Например, не запускать игры, снизить яркость, отключать неиспользуемые сетевые модули, использовать расширенный режим энергосбережения.
Камера
В смартфоне используется 13-мегапиксельный фотомодуль, состоящий из 5 линз. Дополняется он двухтоновой вспышкой, и быстрым фазовым PDAF автофокусом. Значение апертуры составляет F/2.2. Интерфейс камеры простой и лаконичный, но содержит очень много возможностей. Есть отдельные режимы для съемки портретов, макро, панорам, имеется встроенный QR-сканер и многое другое я описываю в видео ниже. Стоит отметить наличие ручных настроек, для самостоятельного выбора значений экспозиции, ISO и прочего.
Дневные снимки порадуют достаточно высокой детализацией, неплохой цветопередачей, хорошей резкостью кадра. Вот несколько снимков:
Вот несколько снимков:
Смартфон может создавать «гифки» (GIF-файлы), пример которой вы видите ниже.
Снимок на селфи камеру:
Максимальное разрешение записи видео – FullHD. Днем ролики пишутся при 30 к/с, в условиях недостатка освещения показатель падает до 20 к/с. Поддерживается замедленная съемка. Фронтальная камера представляет собой 5-мегапиксельный модуль, со светосилой F/2.0. Программно в камеру напичкали кучу всевозможных фильтров. И все ради получения эффекта гладкой кожи. Поддерживается видеосъемка с разрешением 1080p.
Дисплей
В смартфоне установлен IPS экран с диагональю 5 дюймов. Разрешение картинки – 1280 x 720 точек, плотность пикселей составляет 296 PPI. Изображение гладкое и красивое, цвета приятные, можно самостоятельно отрегулировать цветовую температуру. Максимальная яркость экрана не запредельная (397 кд/м²), но вполне достаточная. Применена технология полного ламинирования GFF: изображение контрастное, а сенсорный слой четко отрабатывает все касания.
Покрыт дисплей защитным 2,5D стеклом с хорошими олеофобными способностями.
Сетевые возможности
В комбинированный слот возможна установка двух карточек Nano-SIM. Причем обе могут работать в сетях 2G, 3G и 4G. Устройство оснащается Bluetooth 4.0 BLE с низким энергопотреблением, а также беспроводным Wi-Fi модулем, способным работать в диапазонах 2.4 либо 5 ГГц. Работу спутниковой навигации могут обеспечивать системы геопозиционирования GPS с поддержкой A-GPS, и ГЛОНАСС. Навигация работает, как положено, впрочем, как и все остальные сетевые коммуникации.
Звук
Основной динамик в недорогих смартфонах от Мейзу часто ругают за посредственное качество звучания. И это несмотря на то, что людей, слушающих музыку через динамики, осталось не так уж много. Здесь установлен самый обычный бюджетный громкоговоритель. Громкости хватает, чтобы услышать звонок будильника, уведомлений или вызова. А для прослушивания музыки необходимо использовать хорошие наушники. Тогда будет и качество звучания соответствующее.
Программная часть
Meizu U10, которые реализуются на внутреннем рынке Китая, поставляются с операционкой YunOS. На момент создания этого обзора уже существует первая глобальная мультиязычная прошивка Flyme 5.2.3.0G, основанная на Android 6.0. Как правило, продавцы и магазины устанавливают глобальные прошивки прежде, чем устройство попадет к нам в руки. Более того, самостоятельная перепрошивка не является проблемой для опытных пользователей.
Оболочка Flyme кроме стандартного функционала Андроид обладает собственным интерфейсом и расширенными настройками. Отличается отсутствием кнопки меню (все иконки располагаются на рабочих столах). В большинстве случаев программное обеспечение от Meizu работает достаточно стабильно. К тому же, Flyme постоянно, дорабатывается, и обновляется по мере появления новых версий ПО.
Индивидуальные особенности
Мейзу впервые в среднебюджетном сегменте использует такое сочетание дизайна, материалов корпуса и сборки.
Плюсы и минусы
Плюсы:
- смотрится дорого, изящно и стильно;
- наличие сканера отпечатков;
- качественный дисплей и достаточный уровень производительности;
- приятная цена, с учетом дизайна;
- неплохой дисплей с хорошим олеофобным покрытием;
- хорошая основная фотокамера и множество возможностей и настроек.

Минусы:
- комбинированный слот, приходится отказываться от дополнительной СИМ-карты или от установки флешки;
- неразборная конструкция и стекло на тыльной части не лучшим образом влияют на ремонтопригодность, и на стоимость работы соответственно.
Кому подойдет смартфон
Meizu U10 – это хороший выбор для людей, которые при бюджете не выше 170 USD рассчитывают получить максимально красивый 5-дюймовый смартфон. Кроме дизайна в список критериев можно включить стабильную работу устройства, свежее и функциональное ПО, приличные дневные снимки, достаточную производительность и хороший дисплей.
Наш отзыв о смартфоне Meizu U10
Выпуск Meizu U10 (и U20) – это своеобразный эксперимент компании: взять уже готовую удачную начинку, и поместить в премиальный корпус. Те, кто следят за новинками, возмущаются, мол, «ай-яй-яй, Мейзу, так поступать нехорошо». Но что плохого в подобном решении? Раньше красивый, качественный дизайн был прерогативой лишь имиджевых и флагманских моделей. Но далеко не все были готовы отдавать свои кровные за дорогую и блестящую высокотехнологичную игрушку. Производители это поняли, поэтому сейчас даже бюджетники дешевле 100 долларов могут выглядеть очень даже прилично. И такая тактика работает.
Но далеко не все были готовы отдавать свои кровные за дорогую и блестящую высокотехнологичную игрушку. Производители это поняли, поэтому сейчас даже бюджетники дешевле 100 долларов могут выглядеть очень даже прилично. И такая тактика работает.
Полевая копия цитогенной FISH-карты пахитена кукурузы Использование классической генетики для иллюстрации генных и хромосомных карт кукурузы. О Maize-10-Maze Это мероприятие открыто для публики и проводится учащимися старших классов летней программы обучения лесному хозяйству и охране природы (FACE) 2007 года и исследовательской фермой FAMU в Куинси, Флорида. Цели проекта О кукурузе Геном кукурузы был недавно выбран для секвенирования (так как геном человека был в проекте генома человека), усилие в 32 миллиона долларов, спонсируемое NSF, USDA и DOE (пресс-релиз).Этот полевой проект является частью большой национальной попытки охарактеризовать архитектуру генома кукурузы. Мы представим некоторые известные и классические мутанты кукурузы, которые изучаются и используются генетиками и селекционерами на протяжении многих десятилетий. Кредиты на проекты Часть пропагандистского проекта Maize-10-Maze , поддерживаемого NSF, из проекта цитогенетического картирования, который в настоящее время находится в стадии реализации. Проекты наставничества по естественным наукам в средней школе с Классные проекты и презентации
FIELD 2004, Mission Road Field, FSU Biology, Таллахасси, Флорида Фотографии поля мутантов с летнего поля 2004 года Примечание об источнике мутантов кукурузы : Летняя образовательная программа по лесному хозяйству и охране природы — это трехнедельная летняя программа, цель которой познакомить чернокожих и представителей других меньшинств с научными дисциплинами, такими как лесное хозяйство и сохранение природных ресурсов, включая генетику растений и смежные дисциплины. В этом году, благодаря специальному гранту NSF, доктор Басс работал с доктором Онокписе и его коллегами над включением проекта Genetics кукурузы из бывшего СССР в летнюю программу FACE.  Эта информационно-пропагандистская программа предназначена для ознакомления учащихся из числа меньшинств с аспектами производства и управления кукурузой (кукурузой), о которых они в противном случае не знали бы, тем самым побуждая учащихся старших классов в округах с низким доходом рассматривать растениеводство как будущую карьеру не только в сфере пищевых продуктов. производства, но также в лесном хозяйстве и управлении природными ресурсами. Эта информационно-пропагандистская программа предназначена для ознакомления учащихся из числа меньшинств с аспектами производства и управления кукурузой (кукурузой), о которых они в противном случае не знали бы, тем самым побуждая учащихся старших классов в округах с низким доходом рассматривать растениеводство как будущую карьеру не только в сфере пищевых продуктов. производства, но также в лесном хозяйстве и управлении природными ресурсами.День мутантов кукурузы 2007 года: Дополнительная информация и фотогалереи ко Дню поля 2007 года на исследовательской ферме FAMU в Куинси, Флорида. Мероприятия ко дню поля Sci-Girls II, лето 2008 г.
|
Хлопья кукурузы 10 фунтов, Бренд: Briess by Label Peelers
Кукурузные хлопья с добавлением сбраживаемых сахаров без добавления цвета, тела или солода. Используется во многих сортах пива. Обычно используется только как 10% от общего количества зерна. Кукуруза придает готовому продукту легкий кукурузный привкус. Прежелатинизированный. Использование желтых кукурузных хлопьев в качестве добавки снижает цвет готового пива без снижения первоначальной плотности. Из желтых кукурузных хлопьев получается пиво с мягким, менее солодовым вкусом. Из желтых кукурузных хлопьев получается более сухое и свежее пиво.
Из желтых кукурузных хлопьев получается пиво с мягким, менее солодовым вкусом. Из желтых кукурузных хлопьев получается более сухое и свежее пиво.
ТИПИЧНЫЙ АНАЛИЗ
- Влажность …………………………………… ……………………… 8,0%
- Экстракт FG на сухой основе …………… …………………………….. 75,0%
- Белковая сухая основа …….. ……………………………………….. 10,0%
- Диастатическая сила oLintner ………………………………. незначительно
- Время преобразования ………………………. ………………….. <10 мин.
- Цвет ……………………………………….. …………. 0,8 oLovibond (Degree Lovibond, Series, 52, ячейка 1⁄2 дюйма)
ИНГРЕДИЕНТЫ
Кукуруза; Может содержать пшеницу
КОШЕРНАЯ СЕРТИФИКАЦИЯ
UMK Pareve
ХРАНЕНИЕ И СРОК ГОДНОСТИ
Лучше всего использовать в течение 6 месяцев с даты изготовления. Хранить в сухом месте при температуре <90 oF.
Хранить в сухом месте при температуре <90 oF.
Обращайтесь осторожно, чтобы не сломать хлопья.
Желтые кукурузные хлопья Brewers
ХАРАКТЕРИСТИКИ / ПРИМЕНЕНИЕ
Желтые кукурузные хлопья Brewers можно использовать в качестве добавки к злакам. Хлопья можно добавлять в затор, не используя предварительно зерноварку. Предварительно желатинизированные хлопья дадут более высокий выход и более беспроблемное заваривание, чем обычные хлопья кукурузы. Кроме того, кукуруза была обезжирена (удалено масло), что позволило устранить прогорклость.
Использование желатинизированных желтых кукурузных хлопьев в качестве добавки снижает цвет готового пива без снижения первоначальной плотности.
Добавить непосредственно в затор вместе с солодом. Не фрезеровать. Может использоваться одно- или многотемпературная инфузия. Время преобразования и время фильтрации будут нормальными.
Предварительно желатинизированные желтые кукурузные хлопья дают пиво с мягким, менее солодовым вкусом.

Предварительно желатинизированные желтые кукурузные хлопья дают более сухое и хрустящее пиво.
Использование до 40% в качестве добавки к зерну в общей засыпке
Последовательность Центромеры 10 высококачественной кукурузы выявляет признаки частых событий рекомбинации
Abstract
Предковые центромеры кукурузы содержат длинные участки тандемно расположенных повторов CentC. Обилие тандемных повторов ДНК и центромерных ретротранспозонов (CR) представляет серьезную проблему для полной сборки центромер с использованием традиционных методов секвенирования.Здесь мы сообщаем о почти полной сборке центромеры 10 1,85 Mb кукурузы из инбредного B73 с использованием технологии PacBio и BAC из проекта эталонного генома. Частота ошибок, оцененная из перекрывающихся последовательностей ВАС, составляет 7 × 10 -6 и 5 × 10 -5 для несовпадений и отступов, соответственно. Количество пробелов в области, охватываемой повторной сборкой, было уменьшено со 140 в эталонном геноме до трех. Три экспрессируемых гена расположены между 92 и 477 kb от предполагаемого предкового кластера CentC, который находится в области наивысшей плотности центромерных повторов.Усовершенствованная сборка увеличила количество полноразмерных CR с 5 до 55 и выявила сегментарную дупликацию в 22,7 т.п. Наш анализ предоставляет доказательства частых событий рекомбинации в виде частичных ретротранспозонов, делеций в ретротранспозонах, химерных ретротранспозонов, сегментных дупликаций, включая повторы CentC более высокого порядка, удаленный мономер CentC, центромер-проксимальные инверсии и вставки митохондриальных последовательностей. Реставрация двухцепочечных разрывов ДНК (DSB) является наиболее вероятным механизмом для этих событий и может быть основным драйвером эволюции и разнообразия центромерных повторов.Во многих рассмотренных здесь случаях репарация DSB, по-видимому, опосредуется микрогомологией, это указывает на то, что тандемные повторы, возможно, эволюционировали, чтобы эффективно восстанавливать частые DSB в центромерах.
Три экспрессируемых гена расположены между 92 и 477 kb от предполагаемого предкового кластера CentC, который находится в области наивысшей плотности центромерных повторов.Усовершенствованная сборка увеличила количество полноразмерных CR с 5 до 55 и выявила сегментарную дупликацию в 22,7 т.п. Наш анализ предоставляет доказательства частых событий рекомбинации в виде частичных ретротранспозонов, делеций в ретротранспозонах, химерных ретротранспозонов, сегментных дупликаций, включая повторы CentC более высокого порядка, удаленный мономер CentC, центромер-проксимальные инверсии и вставки митохондриальных последовательностей. Реставрация двухцепочечных разрывов ДНК (DSB) является наиболее вероятным механизмом для этих событий и может быть основным драйвером эволюции и разнообразия центромерных повторов.Во многих рассмотренных здесь случаях репарация DSB, по-видимому, опосредуется микрогомологией, это указывает на то, что тандемные повторы, возможно, эволюционировали, чтобы эффективно восстанавливать частые DSB в центромерах.
Ключевые слова: эволюция центромер, восстановление повреждений ДНК, потеря ДНК на центромерах, гемицентрическая инверсия, незаконная рекомбинация
Введение
Центромеры необходимы для точного разделения хромосом во время деления клеток у высших организмов и обычно видны как первичные перетяжка на хромосоме.Белковая кинетохора, которая формируется на вершине центромеры, взаимодействует непосредственно с микротрубочками веретена, влияя на движение хромосом во время деления клетки. Высокая частота DSB, образующихся во время митозов внутри и вблизи центромер человеческих и мышиных клеток, несущих дефекты митотического веретена, свидетельствует об индуцированном веретеном сдвиге центромеры (Guerrero et al., 2010). Центромер-проксимальные DSB, которые могут приводить к делеции и рекомбинации, хорошо задокументированы и обнаруживаются как инверсии парацентрических плеч хромосомы (например,g., томат Tanksley et al., 1992), транслокации центрического слияния (Робертсона) (например, человеческий Jacobs, 1981) и слияния вложенных хромосом (например, Brachypodium, Murat et al. , 2010; The International Brachypodium Initiative, 2010). , а также нарушение коллинеарности сорго-риса вблизи центромер (Bowers et al., 2005).
, 2010; The International Brachypodium Initiative, 2010). , а также нарушение коллинеарности сорго-риса вблизи центромер (Bowers et al., 2005).
Центромеры-специфические ретротранспозоны (CRs) и длинные тандемные массивы из 156 nt повторов CentC являются ключевыми компонентами ДНК центромер кукурузы (Jiang et al., 2003).Хотя кукуруза отделилась от риса около 50 миллионов лет назад, CentC похож на CentO риса по длине и последовательности (Lee et al., 2005), что указывает на то, что эти повторы сохраняются на своих соответствующих центромерах в течение очень долгого времени. Тем не менее, у домашней кукурузы уровень CentC ниже, чем у ее диких родственников теозинте (Albert et al., 2010; Hufford et al., 2012; Bilinski et al., 2015).
Центромерные ретротранспозоны (CR) были впервые обнаружены как многочисленные центромерные повторы у сорго и ячменя (Miller et al., 1998; Presting et al., 1998). В настоящее время описано шесть семейств CR-элементов для кукурузы и идентифицированы их ортологи в рисе и других травах (Sharma and Presting, 2008, 2014). CR1, CR2 и CR3 кукурузы обладают способностью нацеливать их вставку на центромеры, но мало что известно о механизме нацеливания, как регулируется ретротранспозиция, или о роли, которую эти элементы играют в функции центромеры. По крайней мере, пять различных подгрупп CR1 (от R1 до R5) возникли в результате геномной рекомбинации (Sharma et al., 2008). Сообщалось о тандемных повторах, происходящих от элементов CR (Sharma et al., 2013), а также о случаях конверсии генов (Shi et al., 2010) и снижении синтении кукурузы и сорго (Wang and Bennetzen, 2012) в центромерах кукурузы, но относительно относительно частоты этих событий имеется мало данных. Высококачественные физические карты одной или нескольких центромер кукурузы будут иметь решающее значение для получения более четкой картины эволюции центромеры, но длина центромерных повторов (7-8 kb для элементов CR и десятки или сотни kb для массивов CentC) вызывает неоптимальные сборки центромеры даже в эталонном геноме высококачественной кукурузы, сконструированном из инбредного B73 (Schnable et al.
CR1, CR2 и CR3 кукурузы обладают способностью нацеливать их вставку на центромеры, но мало что известно о механизме нацеливания, как регулируется ретротранспозиция, или о роли, которую эти элементы играют в функции центромеры. По крайней мере, пять различных подгрупп CR1 (от R1 до R5) возникли в результате геномной рекомбинации (Sharma et al., 2008). Сообщалось о тандемных повторах, происходящих от элементов CR (Sharma et al., 2013), а также о случаях конверсии генов (Shi et al., 2010) и снижении синтении кукурузы и сорго (Wang and Bennetzen, 2012) в центромерах кукурузы, но относительно относительно частоты этих событий имеется мало данных. Высококачественные физические карты одной или нескольких центромер кукурузы будут иметь решающее значение для получения более четкой картины эволюции центромеры, но длина центромерных повторов (7-8 kb для элементов CR и десятки или сотни kb для массивов CentC) вызывает неоптимальные сборки центромеры даже в эталонном геноме высококачественной кукурузы, сконструированном из инбредного B73 (Schnable et al. , 2009; Wolfgruber et al., 2009).
, 2009; Wolfgruber et al., 2009).
Мы повторно секвенировали ВАС проекта референсного генома (Schnable et al., 2009), которые соответствуют активной области центромеры 10 кукурузы (CEN10), как определено связыванием центромер-специфического гистона h4 (cenh4), с использованием технологии PacBio. Сборка этих длинных считываний с помощью программного обеспечения PacBio SMRT позволила закрыть почти все пробелы, покрытые нашей сборкой в CEN10 эталонного генома. Длинные чтения PacBio могут охватывать полные элементы CR и повторы более высокого порядка (HOR) CentC, обеспечивая точную сборку и датирование вставок CR и некоторых HOR CentC.
Улучшенная сборка CEN10 выявила доказательства многочисленных DSB, репарируемых посредством гомологически опосредованной внутрицепочечной рекомбинации. Посредством секвенирования сегмента CEN10 CentC у другого инбредного животного мы идентифицировали опосредованную гомологией рекомбинацию, которая привела к делеции одного мономера CentC и созданию нового варианта CentC. Временные рамки для описанных событий делеции, вставки и инверсии, включая по крайней мере одну гемицентрическую инверсию, которая изменила форму CEN10 в течение последних 16,4 тысяч лет, обеспечиваются датировкой вставок CR и сегментарных дупликаций.
Временные рамки для описанных событий делеции, вставки и инверсии, включая по крайней мере одну гемицентрическую инверсию, которая изменила форму CEN10 в течение последних 16,4 тысяч лет, обеспечиваются датировкой вставок CR и сегментарных дупликаций.
Материалы и методы
Получение и секвенирование BAC
Тринадцать BAC из региона CEN10 (Schnable et al., 2009; Wei et al., 2009; Wolfgruber et al., 2009) были повторно секвенированы с использованием технологии long read PacBio. Один дополнительный BAC, отсутствующий в эталонном геноме (№ 11 в таблице S1 и на рисунке), был идентифицирован в GenBank на основании его конечных последовательностей (образцы {«type»: «entrez-nucleotide», «attrs»: {«text»: «ED551002) .1 «,» term_id «:» 115392458 «,» term_text «:» ED551002.1 «}} ED551002.1 и {» type «:» entrez-нуклеотид «,» attrs «: {» text «:» ED551003.1 «,» term_id «:» 115392459 «,» term_text «:» ED551003.1. «}} ED551003.1), соответствующие фланкирующим ВАС и включенные в нашу сборку, чтобы закрыть разрыв суперконтига на физической карте кукурузы (Soderlund et al. , 2000; Wei et al., 2009). ВАС были секвенированы с использованием обеих одиночных молекул PacBio секвенирование (Eid et al., 2009) и секвенирование парных концов Illumina (Bentley et al., 2008). Данные PacBio и Illumina доступны в NCBI под регистрационным номером исследования SRA SRP068233.
, 2000; Wei et al., 2009). ВАС были секвенированы с использованием обеих одиночных молекул PacBio секвенирование (Eid et al., 2009) и секвенирование парных концов Illumina (Bentley et al., 2008). Данные PacBio и Illumina доступны в NCBI под регистрационным номером исследования SRA SRP068233.
Обзор функций CEN10. (A) Два кластера центромерных повторов, содержащие CentC (C1 и C2) в CEN10-содержащем суперконтиге, были разделены наследственной гемицентрической инверсией и теперь разделены областью 2,6 Mb, включающей кластер CR без CentC (NC). CR3 / CentA означает CR3 и CentA (неавтономный CR3). (B) Обогащение анти-cenh4 ChIP-seq показывает, что NC и C2 находятся в активной центромере. Область с измененной последовательностью указывается черной горизонтальной полосой. (C) Обозначены области повторно секвенированного CEN10, охватываемого каждым ВАС. (D) Центромерный повторяющийся контент на окно 100 кб. (E) Позиции трех массивов CentC (# 1 и # 2 прерваны ретротранспозонами), трех транскрибированных генов (зондированных FISH), двух митохондриальных вставок и трех оставшихся пробелов в последовательности (черные вертикальные полосы в матрице CentC # 2) . Стрелки указывают ориентацию массивов CentC; стык двух соседних перевернутых массивов отмечен вертикальной чертой.Стрелки указывают направление транскрипции для трех генов. (F) Предполагаемые даты вставки элемента CR, дублирования области 22,7 kb (горизонтальная черная линия, дублирование отмечено смежными серыми сегментами, обозначенными «L» и «R» и разделенными вертикальной черной линией) и семь более высоких -порядковые повторы (HOR) CentC (парные, в основном перекрывающиеся, зеленые стрелки) указаны на оси y. Три CR с внутренней рекомбинацией (включая CR частично в дупликации 22,7 т.п.н.) отмечены рамкой (черным).Самый молодой из датированных CentC HOR ( Рисунок 5 ) обведен кружком, HOR из Рисунок 6 заключен в рамку. (G) Положения рекомбинантных последовательностей CR, включая три одиночных LTR CR1 ( * = два одиночных элемента, разделенных всего на 2,3 т.п.н.), три химерных элемента (полные, но с несовпадающими TSD, пары LTR заключены в рамку) и 14 частичных элементов CR .
Стрелки указывают ориентацию массивов CentC; стык двух соседних перевернутых массивов отмечен вертикальной чертой.Стрелки указывают направление транскрипции для трех генов. (F) Предполагаемые даты вставки элемента CR, дублирования области 22,7 kb (горизонтальная черная линия, дублирование отмечено смежными серыми сегментами, обозначенными «L» и «R» и разделенными вертикальной черной линией) и семь более высоких -порядковые повторы (HOR) CentC (парные, в основном перекрывающиеся, зеленые стрелки) указаны на оси y. Три CR с внутренней рекомбинацией (включая CR частично в дупликации 22,7 т.п.н.) отмечены рамкой (черным).Самый молодой из датированных CentC HOR ( Рисунок 5 ) обведен кружком, HOR из Рисунок 6 заключен в рамку. (G) Положения рекомбинантных последовательностей CR, включая три одиночных LTR CR1 ( * = два одиночных элемента, разделенных всего на 2,3 т.п.н.), три химерных элемента (полные, но с несовпадающими TSD, пары LTR заключены в рамку) и 14 частичных элементов CR . Для ясности показаны только два ближайших конца химерного элемента, расположенные в дупликации 22,7 т.п.н. Частичный CR1, который может быть искусственно усечен вектором BAC (# 12), показан в позиции 1.63 Мб. (H) Маркеры синтении, общие с хромосомами 7 (SyMAP 7) и 9 (SyMAP 9) сорго, указаны их координатами сорго и кукурузы с ориентацией блоков (линий), обозначенной стрелками. Самый нижний кластер маркеров второго блока синтении (на 1 Mb) инвертирован относительно сорго, но коллинеарен с Brachypodium и рисом. 1.85 Mb CEN10 состоит из четырех разных синтенических сегментов из двух разных хромосом сорго, обозначенных здесь метками, как описано на , рис. 7, и дополнительном рисунке S10.
Для ясности показаны только два ближайших конца химерного элемента, расположенные в дупликации 22,7 т.п.н. Частичный CR1, который может быть искусственно усечен вектором BAC (# 12), показан в позиции 1.63 Мб. (H) Маркеры синтении, общие с хромосомами 7 (SyMAP 7) и 9 (SyMAP 9) сорго, указаны их координатами сорго и кукурузы с ориентацией блоков (линий), обозначенной стрелками. Самый нижний кластер маркеров второго блока синтении (на 1 Mb) инвертирован относительно сорго, но коллинеарен с Brachypodium и рисом. 1.85 Mb CEN10 состоит из четырех разных синтенических сегментов из двух разных хромосом сорго, обозначенных здесь метками, как описано на , рис. 7, и дополнительном рисунке S10.
Для получения ДНК клетки, несущие ВАС, выращивали в среде LB с добавлением 12,5 мкг / мл хлорамфеникола. ДНК ВАС выделяли с использованием набора QIAGEN Large-Construct Kit (QIAGEN Sciences, Inc., Джермантаун, Мэриленд, США). Для секвенирования PacBio было выделено 6–16 мкг ДНК ВАС. Образцы секвенировали с использованием химии XL-C2 и загрузки MagBead с помощью секвенатора PacBio RS II (Pacific Biosciences of California, Inc., Менло-Парк, Калифорния, США) в Центре генома Университета Калифорнии в Дэвисе (Дэвис, Калифорния, США).Использовались ячейки SMRT версий с 1.3.1 по 2.0.0. Для приготовления библиотеки Illumina 5 мкг ВАС ДНК фрагментировали с использованием фермента NEBNext dsDNA Fragmentase (New England Biolabs, Ipswich, Massachusetts, USA) для получения фрагментов с максимальным размером 300–400 п.н. Фрагменты размером 200–300 п.н. очищали в геле с использованием набора для очистки QIAGEN Gel с последующей репарацией концов ДНК с использованием модуля NEBNext End Repair Module и dA-хвостом с использованием NEBNext dA-Tailing. Очистку ДНК после репарации концов и образования dA-хвостов проводили с использованием реагента Agencourt AMPure XP (Beckman Coulter, Inc., Бри, Калифорния, США). К каждому ВАС эквимолярные запасы универсального адаптера и адаптера с уникальным индексом лигировали с использованием лигазы Enzymatics (Enzymatics, Inc.
Образцы секвенировали с использованием химии XL-C2 и загрузки MagBead с помощью секвенатора PacBio RS II (Pacific Biosciences of California, Inc., Менло-Парк, Калифорния, США) в Центре генома Университета Калифорнии в Дэвисе (Дэвис, Калифорния, США).Использовались ячейки SMRT версий с 1.3.1 по 2.0.0. Для приготовления библиотеки Illumina 5 мкг ВАС ДНК фрагментировали с использованием фермента NEBNext dsDNA Fragmentase (New England Biolabs, Ipswich, Massachusetts, USA) для получения фрагментов с максимальным размером 300–400 п.н. Фрагменты размером 200–300 п.н. очищали в геле с использованием набора для очистки QIAGEN Gel с последующей репарацией концов ДНК с использованием модуля NEBNext End Repair Module и dA-хвостом с использованием NEBNext dA-Tailing. Очистку ДНК после репарации концов и образования dA-хвостов проводили с использованием реагента Agencourt AMPure XP (Beckman Coulter, Inc., Бри, Калифорния, США). К каждому ВАС эквимолярные запасы универсального адаптера и адаптера с уникальным индексом лигировали с использованием лигазы Enzymatics (Enzymatics, Inc. , Беверли, Массачусетс, США) в соответствии со стандартными протоколами, а затем реакцию обрабатывали протеиназой К. Реакцию лигирования проводили на гель агарозы и фрагменты размером 400–500 п.н. очищали с помощью набора для очистки QIAGEN Gel. Один цикл ПЦР был проведен с прямым и обратным праймерами Illumina, а очистка ПЦР была проведена с использованием гранул AMPure.Количественную оценку библиотеки проводили с использованием стандартов qPCR от Kapa Biosystems (Уилмингтон, Массачусетс, США), а целостность образцов определяли с помощью Bioanalyzer (Agilent Technologies, Inc., Санта-Клара, Калифорния, США). Секвенирование Illumina MiSeq (Illumina, Inc., Сан-Диего, Калифорния, США) было выполнено в Базовом центре эволюционной генетики Гавайского института морской биологии (Канеохе, Гавайи, США).
, Беверли, Массачусетс, США) в соответствии со стандартными протоколами, а затем реакцию обрабатывали протеиназой К. Реакцию лигирования проводили на гель агарозы и фрагменты размером 400–500 п.н. очищали с помощью набора для очистки QIAGEN Gel. Один цикл ПЦР был проведен с прямым и обратным праймерами Illumina, а очистка ПЦР была проведена с использованием гранул AMPure.Количественную оценку библиотеки проводили с использованием стандартов qPCR от Kapa Biosystems (Уилмингтон, Массачусетс, США), а целостность образцов определяли с помощью Bioanalyzer (Agilent Technologies, Inc., Санта-Клара, Калифорния, США). Секвенирование Illumina MiSeq (Illumina, Inc., Сан-Диего, Калифорния, США) было выполнено в Базовом центре эволюционной генетики Гавайского института морской биологии (Канеохе, Гавайи, США).
Сборка считывателей PacBio и контроль качества
BAC были собраны путем загрузки данных ячеек PacBio SMRT в программное обеспечение SMRT Analysis версии 2.3.0 (https://github. com/PacificBiosciences/SMRT-Analysis) и с использованием протокола HGAP2 (Chin et al., 2013). Пользовательские настройки для минимальной длины субчитания, минимальной длины чтения начального числа и предполагаемого размера вставки («генома») для каждого BAC описаны в таблице S1. Полученная в результате сборка HGAP2 затем была запущена через протокол повторного секвенирования анализа SMRT с использованием однозначно сопоставленных чтений любого размера для генерации окончательного консенсуса по вставке.
com/PacificBiosciences/SMRT-Analysis) и с использованием протокола HGAP2 (Chin et al., 2013). Пользовательские настройки для минимальной длины субчитания, минимальной длины чтения начального числа и предполагаемого размера вставки («генома») для каждого BAC описаны в таблице S1. Полученная в результате сборка HGAP2 затем была запущена через протокол повторного секвенирования анализа SMRT с использованием однозначно сопоставленных чтений любого размера для генерации окончательного консенсуса по вставке.
Перекрытия между вставками ВАС были идентифицированы с помощью BLAST (Camacho et al., 2009) и удален. Разрывы были вставлены в массив CentC из BAC # 11, не охваченный одним считыванием PacBio (позиция 1580,181–1,580,281 нт), между неперекрывающимися BAC №11 и №12 (позиция 1,610,075–1610,175 нт) и внутри кластера CentC в ВАС № 12 при разрыве идентичности с ВАС № 13 (положение 1 617 643–1 627 252 нуклеотида), вызванном делецией в повторно секвенированном ВАС № 12. Сборка CEN10 (лист данных S1 и доступ к GenBank {«type»: «entrez-нуклеотид», «attrs»: {«text»: «KT989678», «term_id»: «963560154», «term_text»: «KT989678»} } KT989678) затем интегрировали в эталонный геном (B73 RefGen_v3 через ftp: // ftp. ensemblgenomes.org/pub/plants/release-18/fasta/zea_mays/dna/Zea_mays.AGPv3.18.dna.*.gz; Schnable et al., 2009) путем замены оригинальной области RefGen_v3 хромосомы 10 из 50,003,470–51,845,973 нуклеотидов на повторную сборку 1,852,772 нуклеотидов. Области {«type»: «entrez-нуклеотид», «attrs»: {«text»: «AC209849.4», «term_id»: «194354100», «term_text»: «AC209849.4»}} AC209849. 4 (BAC № 12), отсутствующие в повторно секвенированном BAC № 12, были идентифицированы BLAST2seq, проверены визуально с использованием изображений JunctionViewer (JV) и извлечены в отдельный файл FASTA (лист данных S2).
ensemblgenomes.org/pub/plants/release-18/fasta/zea_mays/dna/Zea_mays.AGPv3.18.dna.*.gz; Schnable et al., 2009) путем замены оригинальной области RefGen_v3 хромосомы 10 из 50,003,470–51,845,973 нуклеотидов на повторную сборку 1,852,772 нуклеотидов. Области {«type»: «entrez-нуклеотид», «attrs»: {«text»: «AC209849.4», «term_id»: «194354100», «term_text»: «AC209849.4»}} AC209849. 4 (BAC № 12), отсутствующие в повторно секвенированном BAC № 12, были идентифицированы BLAST2seq, проверены визуально с использованием изображений JunctionViewer (JV) и извлечены в отдельный файл FASTA (лист данных S2).
Последовательности ретротранспозона Cinful в BACs № 11–13 были детально исследованы с использованием последовательности Сэнгера из GenBank (где доступно) и данных Illumina и PacBio из усилий по повторному секвенированию, чтобы проверить SNP, которые предполагают сегментарную дупликацию. Пары чтения Illumina (параметры «-X 600 — без смешивания — без дискордантности — без ласточкин хвост») были сопоставлены со сборками PacBio каждого BAC с помощью Bowtie 2 (Langmead and Salzberg, 2012), и был достигнут консенсус. полученные с помощью Integrative Genomics Viewer (Robinson et al., 2011). Согласованные последовательности из считываний Сэнгера (доступные для ВАС № 12–13 из проекта эталонного генома кукурузы) были сгенерированы с использованием программы Geneious (Kearse et al., 2012) для сопоставления считываний с каждой эталонной последовательностью сборки PacBio (средняя чувствительность). Элементы Cinful были извлечены из каждого консенсуса и выровнены с помощью MUSCLE (Эдгар, 2004).
полученные с помощью Integrative Genomics Viewer (Robinson et al., 2011). Согласованные последовательности из считываний Сэнгера (доступные для ВАС № 12–13 из проекта эталонного генома кукурузы) были сгенерированы с использованием программы Geneious (Kearse et al., 2012) для сопоставления считываний с каждой эталонной последовательностью сборки PacBio (средняя чувствительность). Элементы Cinful были извлечены из каждого консенсуса и выровнены с помощью MUSCLE (Эдгар, 2004).
Содержание CR и CentC было рассчитано для каждой собранной вставки ВАС, и показания Illumina, сгенерированные из каждой ВАС, с использованием RepeatMasker с rmblast (repeatmasker.org) и согласованные последовательности, также используемые для аннотаций JV (Wolfgruber and Presting, 2010).
Флуоресценция in situ гибридизацию (FISH) проводили, как описано ранее (Kato et al., 2006; Lamb et al., 2007a), с использованием последовательностей из сборки CEN10. Генные зонды были созданы с помощью ПЦР-амплификации геномной ДНК B73 с использованием праймеров, которые амплифицируют фрагменты, соответствующие положениям 991,393–999,754 нуклеотидов (ген 1), 1,331,762–1,340,808 нуклеотидов (ген 2) и 1,771,713–1,780,104 нуклеотидов (ген 3).
Датирование вставок ретротранспозонов и дупликаций генома
Были датированы LTR ретротранспозоны, вставленные в CEN10-содержащий суперконтиг (позиции исходного эталонного генома (v3) 44 645 284–60 809 161 нуклеотид). RepeatMasker с rmblast использовался для идентификации LTR CR с использованием последовательностей LTR CR1, CR2, CR3 и CentA, используемых для аннотаций JV, и LTR без CR с использованием последовательностей, аннотированных как «LTR» из подмножества кукурузы в GIRI Repbase (Jurka et al. , 2005). Не-CR LTR были идентифицированы только из последовательности, еще не идентифицированной как CR LTR.Сроки были рассчитаны для пар LTR с идентичными 5-нуклеотидными дупликациями фланкирующих целевых сайтов (TSD) и только тогда, когда по крайней мере 2 нуклеотида на краях LTR имели совпадения обратного комплемента, например, 5′-TG с CA-3 ‘. Кроме того, LTR должны принадлежать к одному и тому же подсемейству, например, CR1, и ориентации, а меньший LTR может составлять не менее 90% от более длинной длины LTR. Элементы CR также были датированы с использованием аннотаций JV в качестве ориентира (рисунок S1), что позволяет допускать единичное несоответствие / несоответствие между TSD.
Элементы CR также были датированы с использованием аннотаций JV в качестве ориентира (рисунок S1), что позволяет допускать единичное несоответствие / несоответствие между TSD.
Время вставки оценивали путем выравнивания пар LTR с использованием MUSCLE (Эдгар, 2004) и вычисления значения 2-параметра Кимуры (K2p) (Kimura, 1980) на основе полученного выравнивания с использованием BioPerl (http: // www.bioperl.org/wiki/Main_Page). Значение K2p было переведено в годы с использованием ранее определенной скорости замещения 3,3 × 10 -8 замен / сайт / год (Clark et al., 2005).
Создание и картирование считываний ChIP-Seq анти-cenh4
Поликлональные кроличьи антитела были получены (Cocalico Biologicals Inc., Reamstown, PA, USA) из очищенных 58 N-концевых аминокислот (1–58) белка cenh4 кукурузы продуцируется в Escherichia coli после клонирования в pET19 с N-концевой меткой 6xHis.Белок очищали на никелевой колонке по стандартному протоколу. Перед использованием сыворотку антител очищали аффинной очисткой на колонке, связанной с антигеном.
Незрелые початки (длиной около 7 см) инбредной линии кукурузы сорта В73 использовали для иммунопреципитации хроматина (ChIP). ChIP эксперименты проводились по ранее опубликованным протоколам с некоторыми изменениями. Вкратце, растительный материал измельчали до мелкого порошка в жидком азоте, используя ступку и пестик. Порошок сшивали 1% формальдегидом в сшивающем буфере (0.4 М сахароза; 10 мМ трис-HCl, pH 8,0; 1 мМ ЭДТА; 1 мМ PMSF) в течение 20 минут на льду, и сшивание останавливали добавлением 0,1 М глицина (конечная концентрация) в течение еще 5 минут на льду. После фильтрации через два слоя мираклота неочищенные ядра выделяли с использованием M1 (11,9% гексиленгликоль; 10 мМ KPO4, pH 7,0; 100 мМ NaCl; 5 мМ бета-меркаптоэтанол; 0,1 мМ PMSF, коктейль ингибиторов растительных протеаз) и буфера M2. (8,85% гексиленгликоль; 10 мМ KPO4, pH 7,0; 10 мМ MgCl2; 0,5% Triton X-100; 5 мМ бета-меркаптоэтанол; 100 мМ NaCl).Хроматин в препарате неочищенных ядер расщепляли микрококковой нуклеазой (MNase) в буфере MNB (50 мМ Tris-Cl, pH 8,0; 1 мМ CaCl2; 4 мМ MgCl2; 0,3 M сахароза) при 37 ° C с получением моно- и олигонуклеотидов. нуклеосомы. После очистки с помощью протеина A dynabeads (Invitrogen / Thermo Fisher Scientific, Waltham, MA, USA; каталожный номер 100-02D) хроматин инкубировали с очищенным антителом против ( Zea mays ) cenh4. Антитело кролика IgG было включено в качестве отрицательного контроля. После инкубации в течение ночи при вращении в холодной комнате с температурой 1 ° C комплекс антитело-хроматин иммуноосаждался динабусами протеина А с последующей промывкой, элюированием, обратным перекрестным связыванием и очисткой ДНК.После подтверждения качества ChIP с помощью qPCR, 10 нг ChIPed ДНК использовали для 101 цикла секвенирования парных концов Illumina (Университет Юты, Солт-Лейк-Сити, Юта, США). Входная (хроматиновая) ДНК была вырезана из агарозного геля и секвенирована. Данные Cenh4 ChIP-seq для инбредного B73, а также мононуклеосомная фракция входной ДНК, расщепленной MNase, были депонированы в NCBI под регистрационным номером SRP067358 исследования SRA.
нуклеосомы. После очистки с помощью протеина A dynabeads (Invitrogen / Thermo Fisher Scientific, Waltham, MA, USA; каталожный номер 100-02D) хроматин инкубировали с очищенным антителом против ( Zea mays ) cenh4. Антитело кролика IgG было включено в качестве отрицательного контроля. После инкубации в течение ночи при вращении в холодной комнате с температурой 1 ° C комплекс антитело-хроматин иммуноосаждался динабусами протеина А с последующей промывкой, элюированием, обратным перекрестным связыванием и очисткой ДНК.После подтверждения качества ChIP с помощью qPCR, 10 нг ChIPed ДНК использовали для 101 цикла секвенирования парных концов Illumina (Университет Юты, Солт-Лейк-Сити, Юта, США). Входная (хроматиновая) ДНК была вырезана из агарозного геля и секвенирована. Данные Cenh4 ChIP-seq для инбредного B73, а также мононуклеосомная фракция входной ДНК, расщепленной MNase, были депонированы в NCBI под регистрационным номером SRP067358 исследования SRA.
Входные пары и пары чтения анти-cenh4 ChIP-seq Illumina были сопоставлены с RefGen_v3 с пересмотренным CEN10 (включая все эталонные хромосомные, митохондриальные и пластидные последовательности ДНК) с использованием Bowtie 2 (Langmead and Salzberg, 2012) (параметры «-X 1000» — без смешанного — без диссонанса — без ласточкин хвост »). Оба чтения должны точно соответствовать эталону, и по крайней мере одно должно однозначно картироваться в геноме. Обогащение и охват входом или чтением ChIP-seq определяли с помощью samtools (Li et al., 2009). Покрытие нуклеотидов суммировали для каждого окна размером 100 т.п.н., перекрывающегося на 10 т.п.н. Затем было вычислено среднее значение по 9 окнам и нормализовано к количеству считанных пар в соответствующем наборе данных. Обогащение рассчитывали с шагом 10 т.п.н. по геному путем деления ChIP-seq на вход.
Оба чтения должны точно соответствовать эталону, и по крайней мере одно должно однозначно картироваться в геноме. Обогащение и охват входом или чтением ChIP-seq определяли с помощью samtools (Li et al., 2009). Покрытие нуклеотидов суммировали для каждого окна размером 100 т.п.н., перекрывающегося на 10 т.п.н. Затем было вычислено среднее значение по 9 окнам и нормализовано к количеству считанных пар в соответствующем наборе данных. Обогащение рассчитывали с шагом 10 т.п.н. по геному путем деления ChIP-seq на вход.
Создание образа JV, охватывающего CEN10
Был создан образ JV повторно собранной CEN10 (рисунок S1).Аннотации JV: CR2 LTR красный, CR1 LTR синий, CR CDS коричневый, CR3 LTR розовый, CentA LTR оранжевый, CentC зеленый, не-CR повтор или CR2 UTR серый, митохондриальная ДНК фиолетовый и экспрессируемая генная последовательность риса желтый. Аннотации, полученные с помощью cross_match (прямоугольники), отображаются над результатами BLAST (ориентация указана стрелками)._silver_2.jpg) Все датированные элементы или одиночные LTR были помечены над их аннотациями LTR (номера в рамке), с положительными и отрицательными числами, указывающими дату вставки в тысячах лет и одиночные LTR, соответственно.Показаны последовательности на концах LTR. MUMmer (Kurtz et al., 2004) минимальные длины совпадений ≥ 20 нт показаны внизу изображения, оттянутые самые длинные совпадения к более коротким совпадениям ограничены тем, что уместится в пространстве.
Все датированные элементы или одиночные LTR были помечены над их аннотациями LTR (номера в рамке), с положительными и отрицательными числами, указывающими дату вставки в тысячах лет и одиночные LTR, соответственно.Показаны последовательности на концах LTR. MUMmer (Kurtz et al., 2004) минимальные длины совпадений ≥ 20 нт показаны внизу изображения, оттянутые самые длинные совпадения к более коротким совпадениям ограничены тем, что уместится в пространстве.
Анализ рекомбинированных последовательностей CR
Таблица, описывающая последовательности CR в CEN10, была создана для каждой последовательности CR, имеющей перекрывающиеся аннотации cross_match и BLAST CR гомологии в JV-изображении CEN10 (фиг. S1). Последовательности CR с множеством BLAST HSP в их кодирующих последовательностях (CDS) исследовали на предмет рекомбинации на уровне нуклеотидов.Эти потенциальные рекомбинанты были извлечены и сопоставлены со всеми непрерывными / полными элементами (без CR или других вставок последовательностей и без делеций) одного и того же типа (CR1 или CR2) в CEN10 с использованием MUSCLE. Полученные выравнивания последовательностей визуализировали с помощью UGENE (Okonechnikov et al., 2012) для определения характеристик делеции и вставки.
Полученные выравнивания последовательностей визуализировали с помощью UGENE (Okonechnikov et al., 2012) для определения характеристик делеции и вставки.
Характеристика CentC HOR
Полноразмерные мономеры CentC были идентифицированы в повторно собранных последовательностях с высокой степенью достоверности между положениями 1,476,916–1,564,121 нт и массивом CentC # 3 повторной сборки.Полноразмерные мономеры были идентифицированы как совпадающие с ≥147 нт консенсусного CentC (таблица данных S3) с использованием BLASTN 2.0 (WU-BLAST; http://blast.wustl.edu/). Эти мономеры были пронумерованы в порядке от 5 ‘до 3’ относительно сборки CEN10. Множественное выравнивание последовательностей мономеров было построено с помощью MUSCLE, а самонастраиваемое (1000) дерево соединения соседей было создано с использованием MEGA (Tamura et al., 2013). CentC HOR были идентифицированы с помощью дерева. Три самых длинных HOR (содержащихся в массивах №1–2 на рисунке), а также самый длинный HOR в самом маленьком массиве CentC (массив №3 на рисунке) были датированы путем соединения внутренних полноразмерных мономеров CentC из каждой HOR, выравнивая их с помощью MUSCLE и генерирования даты из совмещения (см. Датирование вставок ретротранспозонов и дубликатов генома).
Датирование вставок ретротранспозонов и дубликатов генома).
Клонирование и анализ фрагмента CentC в сортах B73 и Mo17
Эквивалентные фрагменты CentC из CEN10 инбредов кукурузы B73 и Mo17 были клонированы в высокостабильный линейный вектор pJAZZ (Godiska et al., 2010). Фрагмент Mo17 был клонирован непосредственно из цельной геномной ДНК без ПЦР-амплификации (таблица данных S4 и доступ в GenBank {«type»: «entrez-nucleotide», «attrs»: {«text»: «KT989679», «term_id»: «952988122) «,» term_text «:» KT989679 «}} KT989679), а фрагмент B73 был субклонирован с использованием BAC (# 13 ZMMBBb-410L22 в таблице S1) в пути мозаики CEN10.Ткань молодого сорта кукурузы Mo17 растирали в жидком азоте и инкубировали в буфере для экстракции ДНК перед экстракцией ДНК с использованием хлороформа, а затем осаждением с использованием этанола. Затем осадок расщепляли в течение ночи при 37 ° C с помощью Hae III, снова осаждали и ремонтировали концы с использованием набора для ремонта концов Lucigen DNATerminator End Repair Kit (Lucigen Corp. , Миддлтон, Висконсин, США). Концевую репарированную ДНК в растворе пропускали через 0,6% агарозный гель и визуализировали с помощью SYBR Safe DNA Gel Stain (Life Technologies, Grand Island, NY, USA) в синем свете.Полосы ≥8 т.п.н. из лунок геля вырезали и очищали с использованием набора для экстракции геля Epoch (Epoch Life Science, Missouri City, TX, USA). Осажденную этанолом ДНК из гелевого экстракта затем лигировали в векторы pJAZZ с использованием набора для линейного клонирования BigEasy v2.0 (Lucigen Corp.) и трансформировали в E.coli , используя процедуру, описанную в BigEasy Kit. Инкубированные трансформанты высевали на YT-агар канамицина с X-gal и IPTG. Белые колонии собирали в воду и повторно взяли, затем подвергали скринингу ПЦР с использованием векторных праймеров (SL1 5′-CAGTCCAGTTACGCTGGAGTC-3 ‘и NZRevC 5′-AAATGGTCAGTTAATCAGTTCT-3′) и праймеров CentC (CentC_F 5′-TCCAAAACTCGATGT’T и CentC_F 5’-TCCAAAACTCGATGT ‘ -GTGGATTGGGGCATGTTCG-3 ′).Продукты ПЦР пропускали через 2% агарозный гель для идентификации полос мономеров / димеров / тримеров 156-нуклеотидного повтора CentC.
, Миддлтон, Висконсин, США). Концевую репарированную ДНК в растворе пропускали через 0,6% агарозный гель и визуализировали с помощью SYBR Safe DNA Gel Stain (Life Technologies, Grand Island, NY, USA) в синем свете.Полосы ≥8 т.п.н. из лунок геля вырезали и очищали с использованием набора для экстракции геля Epoch (Epoch Life Science, Missouri City, TX, USA). Осажденную этанолом ДНК из гелевого экстракта затем лигировали в векторы pJAZZ с использованием набора для линейного клонирования BigEasy v2.0 (Lucigen Corp.) и трансформировали в E.coli , используя процедуру, описанную в BigEasy Kit. Инкубированные трансформанты высевали на YT-агар канамицина с X-gal и IPTG. Белые колонии собирали в воду и повторно взяли, затем подвергали скринингу ПЦР с использованием векторных праймеров (SL1 5′-CAGTCCAGTTACGCTGGAGTC-3 ‘и NZRevC 5′-AAATGGTCAGTTAATCAGTTCT-3′) и праймеров CentC (CentC_F 5′-TCCAAAACTCGATGT’T и CentC_F 5’-TCCAAAACTCGATGT ‘ -GTGGATTGGGGCATGTTCG-3 ′).Продукты ПЦР пропускали через 2% агарозный гель для идентификации полос мономеров / димеров / тримеров 156-нуклеотидного повтора CentC. Клоны с ожидаемыми размерами полос выращивали в среде TB с канамицином и раствором для индукции арабинозы в течение ночи при 250 об / мин и 37 ° C. Затем плазмиды выделяли с использованием набора QIAGEN Miniprep (QIAGEN Inc., Валенсия, Калифорния, США). Раствор, содержащий вектор, обрабатывали Not I при 37 ° C для высвобождения вставки и обработки на 0,6% агарозном геле. Идентифицировали клон с полосой, содержащей три сильные полосы для двух векторных плеч и одну вставку, и полосу вставки вырезали и очищали с использованием набора MACHEREY-NAGEL NucleoSpin Gel и PCR Clean-up (Bethlehem, PA, USA).Очищенную ДНК обрабатывали ультразвуком в течение 20 с, репарировали по концам, прогоняли на 1% агарозном геле и выделяли полосы размером 1-8 т.п.н., а затем трансформировали в вектор pJAZZ с помощью электропорации. Затем колонии подвергали скринингу на полосы мономера / димера / тримеров CentC с использованием ПЦР и 2% агарозного геля, как описано ранее, затем ДНК из колоний, содержащих CentC, секвенировали по Сэнгеру в Pacific Biosciences Research Center Biotech Core (Гонолулу, Гавайи, США) с использованием вектора праймеры SL1 и NZRevC.
Клоны с ожидаемыми размерами полос выращивали в среде TB с канамицином и раствором для индукции арабинозы в течение ночи при 250 об / мин и 37 ° C. Затем плазмиды выделяли с использованием набора QIAGEN Miniprep (QIAGEN Inc., Валенсия, Калифорния, США). Раствор, содержащий вектор, обрабатывали Not I при 37 ° C для высвобождения вставки и обработки на 0,6% агарозном геле. Идентифицировали клон с полосой, содержащей три сильные полосы для двух векторных плеч и одну вставку, и полосу вставки вырезали и очищали с использованием набора MACHEREY-NAGEL NucleoSpin Gel и PCR Clean-up (Bethlehem, PA, USA).Очищенную ДНК обрабатывали ультразвуком в течение 20 с, репарировали по концам, прогоняли на 1% агарозном геле и выделяли полосы размером 1-8 т.п.н., а затем трансформировали в вектор pJAZZ с помощью электропорации. Затем колонии подвергали скринингу на полосы мономера / димера / тримеров CentC с использованием ПЦР и 2% агарозного геля, как описано ранее, затем ДНК из колоний, содержащих CentC, секвенировали по Сэнгеру в Pacific Biosciences Research Center Biotech Core (Гонолулу, Гавайи, США) с использованием вектора праймеры SL1 и NZRevC. Последовательности Сэнгера (доступные в NCBI Trace Archive TI под номерами 2343263554-2343263871) были собраны в Consed (Gordon et al., 1998), где расхождения между собранными чтениями редактировались вручную перед генерацией согласованной последовательности. ДНК B73 BAC выделяли с использованием набора QIAGEN Large-Construct (QIAGEN Inc.), затем субклонировали в pJAZZ, подвергали скринингу, секвенировали и собирали, как это сделано для ДНК Mo17.
Последовательности Сэнгера (доступные в NCBI Trace Archive TI под номерами 2343263554-2343263871) были собраны в Consed (Gordon et al., 1998), где расхождения между собранными чтениями редактировались вручную перед генерацией согласованной последовательности. ДНК B73 BAC выделяли с использованием набора QIAGEN Large-Construct (QIAGEN Inc.), затем субклонировали в pJAZZ, подвергали скринингу, секвенировали и собирали, как это сделано для ДНК Mo17.
Дату расхождения между последовательностями B73 и Mo17 CentC рассчитывали путем генерации выравнивания MUSCLE их сегментов CentC, вычисления расстояния K2p от выравнивания, а затем вычисления даты по расстоянию K2p, как это было сделано для дат вставки CR.
Полноразмерные мономеры CentC были идентифицированы в массиве B73, HOR были идентифицированы из флогенетического дерева мономеров, а полноразмерные мономеры были объединены и датированы, как описано ранее. HOR были дополнительно переопределены с использованием аннотаций MUMmer (—maxmatch) в JV путем перемещения границ HOR в соответствии с наиболее длинным совпадением последовательности.
Картирование синтенных маркеров сорго
Ожидается, что консервативные однокопийные последовательности в маркерах перицентромер (CSCP) (Wang and Bennetzen, 2012) будут фланкировать предковые центромеры и были сопоставлены с пересмотренным эталонным геномом с использованием MUMmer (сквозной уникальный отображение).Независимо, генные маркеры синтении сорго-кукурузы были идентифицированы с помощью программного обеспечения SyMAP (Soderlund et al., 2011), запущенного на локальном компьютере и сравнивающего измененный эталонный геном кукурузы только с теми последовательностями эталонного генома раннего выпуска версии 2.1 сорго, которые соответствуют аннотированные гены (Paterson et al., 2009). Последовательность и положения генов сорго были получены от Phytozome (http://www.phytozome.net/). Синтенические маркеры были сгруппированы в блоки вручную после визуализации данных.
Определение экспрессии генных последовательностей в CEN10
Последовательности генов в CEN10 были помечены в соответствии с аннотациями RefGen_v3 на MaizeGDB.org (Andorf et al., 2015). Аннотированные последовательности генов на JV-изображении повторно собранного CEN10 (фиг. S1) были переведены в исходные позиции RefGen_v3 с использованием (перекрывающихся генов) маркеров SyMAP. Экспрессию определяли путем сопоставления данных РНК-seq сорта кукурузы B73 (NCBI BioProject, доступ PRJNA219741) к повторно собранной последовательности CEN10 с использованием TopHat (Trapnell et al., 2009).
Идентификация содержания CR и CentC в суперконтиге CEN10
Для идентификации содержания CR и CentC в суперконтиге, содержащем CEN10, был проведен конкурентный BLAST, как описано ранее (Schnable et al., 2009), за исключением того, что 1) CentC был включен с CRs и 2) BLAST использовался вместо WU-BLAST.
Результаты
Ресеквенирование значительно улучшает сборку CEN10
Центромерная область хромосомы 10 кукурузы содержит три кластера центромерных повторов (рисунок): C1 и C2, которые содержат тандемные кластеры CentC, и NC, область с высоким содержанием CR, лишенная CentC.Области CentC C1 и C2 разделены на 2,6 МБ, могут быть визуализированы как два отдельных сигнала FISH (например, https://birchler.biology.missouri.edu/wp-content/uploads/2015/06/166-33_B73.jpg ) и, вероятно, являются результатом гемицентрической инверсии (Lamb et al., 2007b). Активная центромера 10 (CEN10) инбредного B73, т.е. область, покрытая нуклеосомами cenh4 (рисунок), включает NC и C2 и представляет собой неоцентромеру, колонизированную нуклеосомами cenh4 несколько тысяч лет назад (Schneider et al., 2016).
Четырнадцать перекрывающихся ВАС, охватывающих активный CEN10 (рисунок), включая один, выбранный для закрытия известного промежутка между суперконтигами, были секвенированы на большую глубину с использованием технологий PacBio (Таблица S1) и Illumina (Таблица S2).Процент содержания CentC и CR в сборках PacBio BAC отличался от такового в считывателях Illumina всего на 0,1–2,8% (таблица S2) от размера вставки BAC, что указывает на то, что сборки точно отражают повторяющееся содержание каждого BAC. Однако наша собранная вставка ВАС # 12 намного меньше (63 205 нуклеотидов), чем соответствующая последовательность GenBank {«type»: «entrez-nucleotide», «attrs»: {«text»: «AC209849», «term_id»: «684625572 «,» term_text «:» AC209849 «}} AC209849 (164 526 н.) из-за потери значительной части вставки ВАС (включая 75 222 н. CentC и 6 865 н. CR) через CentC-опосредованную рекомбинацию во время размножения (лист данных S2 ).
BAC # 11, который изначально был выбран для закрытия разрыва суперконтига (Wei et al., 2009), заканчивается элементом Cinful. Высокая частота SNP между этим элементом Cinful по сравнению с общим для BAC №12 и №13 (6 SNP в 4,408 нуклеотида, таблица S3) по сравнению с рассчитанной частотой ошибок секвенирования (1 SNP на> 147 kb см. Ниже) предполагает, что эти элементы являются результат недавней сегментарной дупликации. Независимо созданные данные о последовательности Illumina и, при наличии, данные о последовательности Сэнгера подтвердили 6 SNP, обнаруженные в последовательности PacBio, и, таким образом, наличие сегментарной дупликации в этой области.Следовательно, разрыв суперконтига остается в нашей сборке (позиция 1,610,075–1,610,175 нт), и ВАС # 11 не перекрывается с ВАС # 12.
Независимо собранная последовательность из перекрывающихся вставок ВАС (рисунок) выявила частоту ошибок 1 SNP на 147 437 нуклеотидов и 1 отступ на 18 430 нуклеотидов (таблица S4). Большинство инделей (22/24) содержали единственный нуклеотид на концах мононуклеотидных рядов длиной от 5 до 33 нуклеотидов (10,2 ± 5,8 нуклеотидов). Все перекрытия ВАС были объединены для получения последовательности CEN10 размером 1,85 Мбайт (таблица данных S1), которая содержит в общей сложности 237 593 нуклеотида CR1, 177 586 нуклеотидов CR2, 17 630 нуклеотидов CR3 и CentA и 58 169 нуклеотидов CentC.Повторно упорядоченный CEN10 содержит только три пробела, один из которых расположен в большом кластере CentC BAC # 11, который не может быть охвачен чтениями PacBio, другой представляет собой пробел в минимальном пути мозаики между BAC # 11 и # 12, а третий происходит из-за потери последовательности в нашем BAC № 12.
Число правильно собранных элементов CR с совпадающими LTR увеличилось с пяти в эталонном геноме до 42 (до включения последовательностей, внесенных BAC # 11). Последовательность BAC # 11 завершает два CR в BAC # 10 и добавляет 11 дополнительных элементов CR с соответствующими LTR (всего 13).В этой сборке мы исправили пять CR с несовпадающими TSD и пять одиночных LTR, которые были неправильно собраны в эталонном геноме.
Распределение центромерных повторов в CEN10
Два CR-богатых кластера в CEN10 разделены 272 т.п.н. (рисунок). Нисходящий кластер содержит три массива CentC C2 (рисунок), первые два из которых указывают в противоположных направлениях и содержат многочисленные вставки CR, а также третий небольшой (<5 т.п.н.) массив CentC, в котором отсутствуют вставки ретротранспозона.Время вставки элементов CR в два кластера, богатых CR, колеблется от 0 до 650 тыс. Лет назад (рисунок), но нижележащий кластер CR содержит гораздо большее количество одиночных, химерных и частичных элементов CR, чем восходящий кластер NC (рисунок ). Синтенические маркеры убедительно указывают на то, что NC и C2, теперь разделенные несинтенической областью 3.3, когда-то были смежными друг с другом (рисунок). Взятые вместе данные на рис. Предполагают, что C2 маркирует место предков центромеры до того, как одна или несколько гемицентрических инверсий переместили некоторые из старых CR элементов в NC и вставили ранее нецентромерную область 3.3 в CEN10.
Гены в CEN10
Три гена, которые лежат очень близко к предковой центромере, содержащей кластер CentC, были подтверждены FISH (рисунок и рисунок S2). Ген 1 (GRMZM2G137715, экспрессируется в листьях) расположен на 329 т.п.н. выше гена 2 (GRMZM2G361718, экспрессируется в корне), который находится непосредственно рядом с недавно вставленным CR1 и прямо перед наиболее плотным центромерных повторов участком CEN10. Третий ген, созданный путем слияния GRMZM2G101098 с GRMZM5G846522 на основе данных RNA-seq (не показаны) после правильной сборки этой области, находится <100 т.п.н. ниже конечного кластера CentC и высоко экспрессируется в молодых тканях (MaizeGDB.org).
Доказательства частых DSB в CEN10
Доказательства многочисленных DSB, обнаруженных при повторной сборке CEN10 в форме рекомбинированных или удаленных повторяющихся последовательностей, и геномных перестроек относительно генома сорго, перечислены ниже. По возможности, делеции в элементах CR были датированы на основе времени вставки ретротранспозона, а даты дупликаций были датированы на основе расхождения исходной и дублированной последовательностей.
Потерянные и рекомбинированные последовательности CR
Три одиночных LTR (рисунок), семь элементов CR с большими внутренними делециями (два из которых имеют одну и ту же делецию) (рисунок и рисунок S3) и три химерных элемента CR (фланкированные несовпадающими TSD) выявить частые рекомбинации в CEN10.CEN10 содержит 14 частичных последовательностей CR (например, одиночный LTR, соединенный с неполным CDS), за исключением сегментно дублированных частичных последовательностей, одиночных LTR и элемента, усеченного вектором BAC (рисунок и №70 в таблице S5). Частичные элементы, вероятно, являются результатом гомологически опосредованной внутрицепочечной рекомбинации, как видно на недавно вставленном CR2 (идентичные LTR указывают время вставки <16,4 тыс. Лет назад), который имеет внутреннюю делецию 5 т.п.н., ограниченную 11 нуклеотидами идентичной последовательности (рисунок). Другие внутренние делеции CR содержат 1-3 идентичных нуклеотида, фланкирующих делеции (Рисунок S3).
Внутренняя делеция CR в результате гомологически опосредованной рекомбинации . Элемент CR2 (№ 74 в Таблице S5) имеет идентичные LTR, которые датируют вставку и последующую рекомбинацию этого элемента <16,4 тыс. Лет назад. (A) Линейный рисунок иллюстрирует (часть 1) рекомбинацию между UTR (серый) и CDS (коричневый), приводящую к наблюдаемому усеченному CR, идентифицированному в CEN10 (часть 2). CR2 LTR окрашены в красный цвет. (B) Выравнивание соединения делеций UTR и CDS с консенсусными последовательностями CR2 показывает, что делеция опосредуется 11 нуклеотидами идентичной последовательности (подчеркнута).
CR1 # 52.2 содержит то, что, по-видимому, является дупликацией 162 нуклеотидов в его кодирующей последовательности полипротеина, что на самом деле связано с двойной рекомбинацией между вложенными элементами CR1 (рисунок). Одна рекомбинация с участием пяти нуклеотидов с идентичной последовательностью, которые повторяются в полипротеине CR1, объединила нижележащую область одного элемента с вышележащей областью второго элемента, и ее можно было отличить только от локальной дупликации, поскольку эти два элемента принадлежат к разным подтипам.В другом примере два CR1 вставлены и рекомбинированы с третьим CR1 (рис. S3C), образуя два химерных элемента.
Дупликация CR CDS из вложенной вставки и рекомбинации . Два элемента CR1, которые на первый взгляд кажутся простыми вложенными вставками, со временем вставки 326 тыс. Лет назад и 188 тыс. Лет назад для # 52.2 и # 53.2, соответственно (таблица S5). При более внимательном рассмотрении обнаруживается, что элемент # 53.2 (принадлежащий к одной из пяти (с R1 по R5) подгрупп CR1 (Sharma et al., 2008)), вставленный в химерный элемент CR1, который содержит очевидную дупликацию ~ 162 нуклеотидов. Этот химерный элемент является результатом внутрицепочечной рекомбинации между двумя различными 5-нуклеотидными областями элемента R4 и R2, в частности, между нижележащим CR1-R4 и вышележащим CR1-R2 участком, который добавляет 162 нуклеотида к полипротеиновой области этого элемента. (A) Линейные рисунки, показывающие события, ведущие к текущей последовательности: первоначальная вставка CR1-R4 в CR1-R2 ( * указывает, что точное место вставки неизвестно), за которыми следовали две рекомбинации, приводящие к потере LTR R4.Поскольку вторая рекомбинация включала два положения смещения (красный в части 3), она выглядит как дупликация, которую можно идентифицировать только как продукт рекомбинации, потому что вышестоящая, полученная из R4, и нижележащая, производная от R2, копии содержат разные аллели в концы их компакт-дисков. Аннотации JV показаны в части 5 (CR1 LTR синие, CDS желтые, UTR без аннотаций). (B) Выравнивание, показывающее 5 нуклеотидов идентичной последовательности между соединением альтернативного конца и соответствующими областями в R2 и R4.Штриховые рисунки, окрашенные в серый цвет, как аннотации JV, и UTR.
Неполные митохондриальные последовательности
Два фрагментированных кластера митохондриальных последовательностей в CEN10 (рисунок) предоставляют дополнительные доказательства разрыва двухцепочечной ДНК в центромере. Кластеры расположены на расстоянии 115 kb друг от друга и выровнены по двум и трем различным областям митохондриального генома соответственно. Порядок и ориентация этих ядерных митохондриальных фрагментов относительно митохондриального генома кукурузы предполагают, что гомологически опосредованная рекомбинация уменьшала первоначально гораздо большую митохондриальную вставку в эти фрагменты (Рисунок S4).Фактически, исследование соединений, образовавшихся в результате этих делеций, относительно контрольной последовательности митохондрий кукурузы подтвердило, что две из делеций связаны с гомологиями по крайней мере трех идентичных нуклеотидов. Третья делеция могла произойти в результате нескольких событий или представлять область, в которой эталонный митохондриальный геном отличается от вставленной последовательности.
Большая смежная сегментарная дупликация в CEN10
Сегментная дупликация 22,7 т.п.н. (рисунок), которая начинается в CDS CR1 и заканчивается внутри LTR CR1 (рисунок S3D), имеет те же пять нуклеотидов на 5′-конце восходящего и 3 ′ конец нижележащего дублированного сегмента, а также на стыке между ними (рисунок).Наиболее правдоподобным объяснением является разрыв двухцепочечной ДНК с последующей репарацией одноцепочечного альтернативного присоединения концов (McVey and Lee, 2008) в месте соединения дупликации (между идентичными 5 нуклеотидами последовательностей CDS и LTR).
Альтернативное соединение концов при образовании смежной сегментарной дупликации . (A) Обзор дублирования. Фрагмент размером 22,7 т.п.н., состоящий из нескольких вложенных и частичных CR1, одиночного LTR CR2 и геномного повтора (серый), дублировали локально (рисунок S3D).Два полноразмерных элемента CR2 впоследствии вставляются в дупликацию, расположенную ниже по потоку (в рамке). CR1 LTRs синий, CR2 LTRs красный и CDS желто-коричневый. (B) Соответствие 5 нуклеотидов между областью, кодирующей полипротеин, на 5′-конце исходного фрагмента 22,7 т.п.н. (верхняя последовательность) и CR1 LTR (представлен 3′-концом дублированной области, нижняя последовательность), вероятно, опосредовано эта тандемная дупликация путем альтернативного присоединения концов ДНК к репарации после двухцепочечного разрыва ДНК. Аннотации СП: CR1 LTR синие, CDS желтые.
Недавно сформированный CentC HOR является результатом смежной дупликации
Филогенетическое дерево было построено (Рисунок S5) из всех полноразмерных мономеров CentC (Таблица S6) массивов №1 и №3 (Рисунок), а также высококачественная последовательность массива №2 (до позиции 1 564 121 нт) для идентификации HOR. Были рассчитаны даты дублирования для самых длинных HOR в каждом из трех массивов (рисунок), исключая первый и последний мономеры каждого HOR. Кроме того, все четыре HOR области массива # 2, которые были повторно секвенированы с помощью технологии Сэнгера (см. Потеря мономера CentC в результате гомологически опосредованной рекомбинации) у инбредов B73 и Mo17, были датированы (заключены в рамку на рисунке).Семь дат дублирования колеблются от 98 до 498 тыс. Лет назад. Самый молодой срок получается при смежной сегментарной дупликации (рисунок).
Недавно созданный CentC HOR является смежным дубликатом . Самый молодой (98 тыс. Лет назад) из четырех датированных HOR CentC в CEN10 (обведен на рисунке) представляет собой 10-мономерную смежную сегментарную дупликацию. (A) JV — аннотация дублированных левого (L) и правого (R) сегментов HOR с нумерацией мономеров по порядку. Координаты мономера приведены в таблице S6 (2A и 2B).R HOR нарушается CR1 с идентичными LTR (расчетное время вставки 0–25,5 тыс. Лет назад). (B) Мономеры L и R показаны в дереве соединения соседей, чтобы указать, что большинство похожих мономеров находятся в эквивалентных положениях в L и R. Показаны проценты начальной загрузки (1000) в точках ветвления. Шкала представляет собой нуклеотидные различия. Идентичны только пары мономеров 5 и 7 — мономеры 3L и 3R отличаются на один индель. Аннотации СП: CentC зеленый, CR1 LTR синий, CDS желто-коричневый.
Потеря мономера CentC в результате гомологически опосредованной рекомбинации
Кластер CentC на ВАС № 13, состоящий приблизительно из 50 мономеров и фланкированный ретротранспозонами Xilon и Cinful, был полностью секвенирован с использованием технологии Sanger или PacBio.Две полученные консенсусные последовательности были идентичны на 100%. Этот массив CentC содержит 4 HOR, которые дублируются между 131 и 297 тыс. Лет назад (рисунок). Самый молодой и самый старый из четырех HOR являются смежными сегментарными дупликациями (рисунок и рисунок S6). Соответствующий участок был клонирован и секвенирован из инбредного Mo17 кукурузы (таблица данных S4) с использованием методов, оптимизированных для стабилизации тандемных повторов. Сравнение Mo17 с последовательностью B73 выявило делецию одного мономера CentC из последовательности Mo17 (рисунок) посредством рекомбинации между мономерами M11 и M12 (рисунок S7), в результате чего рекомбинантный мономер M11 ‘образовался из 5’-области M11. и 3 ′ конец M12.Этот новый химерный мономер образует собственную кладу по сравнению с другими мономерами в массиве (рис. S8) и может быть уникальным в геноме кукурузы. Дата расхождения 67,9 тыс. Лет назад рассчитана между Mo17 и B73 CentC.
Мономер CentC удален из массива инбредных Mo17 . Отличительный массив CEN10 CentC (между вставками ретротранспозона Xilon и Cinful) инбредного B73 совмещен с его аналогом Mo17. Мономеры (отмечены стрелками) сгруппированы в HOR (тонкие стрелки над мономерами) с использованием филогенетического дерева (рисунок S8).Часть первого черного HOR отсутствует в Mo17, где химерный мономер (M11 ‘) возник в результате делеции мономеров 3’ M11 и 5 ‘M12. Эта делеция опосредована областью из 28 идентичных нуклеотидов, присутствующих в мономерах B73, B11 и B12 (фигура S7). Массив Mo17 CentC включает вставку CR1 в M47, которая нарушает второй красный HOR и отсутствует в массиве B73. Сегмент Mo17 после вставки CR1 в элемент Cinful был подтвержден с помощью ПЦР-амплификации и секвенирования. Одна последовательность CentC ( * ), аналогичная другим коричневым мономерам, содержит 40-нуклеотидную вставку в обоих инбредах.Самый молодой (синий) и самый старый (зеленый) HOR датируются 131 тыс. Лет назад и 297 тыс. Лет назад, соответственно. Потенциально рекомбинантные мономеры на концах HOR были исключены из сопоставления, используемого для датирования событий дупликации.
Хромосомные инверсии изменили форму CEN10
Инверсия, которая разбила исходный кластер CentC на две области, привела к одному неактивному (C1) и одному активному (C2) кластеру CentC. Элементы CR продолжали вставляться только в активный кластер CentC, таким образом, время инверсии можно датировать ~ 350 тыс. Лет назад на основе самой последней вставки CR в C1.Дополнительные инверсии, включая некоторые совсем недавние (<16,4 тыс. Лет назад), переместили последовательности CR к верхней границе текущей центромеры и ранее перицентрической области к сегменту CEN10 между NC и C2 (рисунок и рисунок S9B).
Суперконтиг CEN10 и гемицентрические инверсии. (A) Семь перемешанных блоков маркеров хромосомы 7 синтенного сорго (SyMAP 7) пронумерованы в соответствии с их положением в сорго, направление указано стрелками. В рамке C1 и C2 обозначены положения кластеров CentC, NC обозначает кластер CR без CentC (рисунок).Область связывания cenh4 CEN10 отмечена рамкой, окружающей NC и C2. Показаны единичные копии перицентромерных маркеров, консервативных между сорго и кукурузой (CSCP). Маркеры CSCP 7 хромосомы 7 сорго находятся рядом с C1. Маркеры хромосомы 9 сорго (SyMAP 9), которые происходили от конца более крупного синтенического блока на расстоянии 40 Мб (Рисунок S10), были депонированы рядом с CEN10 кукурузы путем нескольких инверсий с участием центромер-проксимальных DSB, что подтверждается присутствием элемента CR1 на дальнем конце блока SyMAP 9 (рисунок S9B).Концы блоков не совпадают ни с одним из двух разрывов суперконтигов в этой области эталонного генома, что указывает на то, что разрывы в синтении являются реальными, а не артефактами сборки последовательности. (B) CR и не-CR элементы, графически отображенные по расчетному времени вставки и положению хромосомы, показывают, что C1 стал неактивным> 300 тысяч лет назад. Элемент CR, использованный для определения даты отделения C1 от C2, приводящего к инактивации C1, обведен кружком. CR3 / CentA = CR3 и CentA (неавтономный CR3).
В дополнение к гемицентрической инверсии, которая разбивает исходный кластер CentC на C1 и C2, ряд других инверсий может быть реконструирован на основе микросинтении сорго.Повторная сборка CEN10 содержит маркеры синтении, которые разнесены на расстояние до 9 МБ и не примыкают друг к другу в хромосоме 7 сорго (рисунок). Эти маркеры являются членами более крупных синтенических блоков, охватывающих ~ 45 Мб в хромосоме 10 кукурузы (Рисунок S10). CEN10 включает концы синтенных блоков 2 и 3 (из 4) хромосомы сорго 7 и маркеры с конца блока 9 синтенной хромосомы сорго, который находится на ~ 40 мб ниже по течению. Эти остатки более крупных синтенических блоков были перемещены в CEN10 посредством множественных инверсий. Синтенические блоки 2 и 3 сильно зашифрованы (подразделы 3.1, 2.1, 3.3, 2.2, 3.2 и 2.3 на рисунке) относительно их положения в сорго, что указывает на то, что в ходе эволюции CEN10 кукурузы произошло несколько дополнительных инверсий. В целом, по крайней мере 8 и, возможно, 17 инверсий (при условии, что вставки CR следуют за активной центромерой) необходимы для объяснения текущего расположения синтенных маркеров сорго внутри и вокруг CEN10 (Рисунок S9).
Обсуждение
Высококачественная последовательность полной центромеры кукурузы представляет собой значительный шаг вперед по сравнению с текущим сильно фрагментированным эталонным геномом, состоящим из неупорядоченных и неориентированных контигов последовательностей, и позволяет детально изучить центромерные повторы и геномные перестройки в центромере.Пакеты PacBio, охватывающие ретротранспозоны, обеспечивают уверенность в отношении обеих последовательностей TSD для каждого элемента CR, что помогает распутать сложные перестройки, которые мы документируем для элементов в CEN10, и разрешить большие тандемные дублирования. Более того, эта новая последовательность обеспечивает определенность порядка и ориентации синтенических маркеров и, таким образом, реконструкцию недавних геномных перестроек. Полное геномное секвенирование с более длинными считываниями более высокого качества в сочетании с другими новыми технологиями физического картирования может позволить закрыть оставшиеся пробелы в будущем.
Полная последовательность центромеры подобной длины и качества доступна только для CEN8 риса (Nagaki et al., 2004; Wu et al., 2004, 2009), который также характеризуется массивами инвертированных тандемных повторов, многочисленными элементами CR и наличие активных генов. Кроме того, сегментарные дупликации и многочисленные центромеры-проксимальные инверсии различают CEN8 разных видов или подвидов риса (Ma and Bennetzen, 2006; Ma and Jackson, 2006; Ma et al., 2007), что указывает на то, что эти события не специфичны для недавно полиплоидизированных геном кукурузы с его большими хромосомами.
Эффективная репарация DSB — основная сила в эволюции повторов центромер
Сравнение небольшого массива CentC в CEN10, который фланкирован ретротранспозоном Xilon и Cinful и был секвенирован с высоким качеством в инбредных генах B73 и Mo17, выявило вставку a Элемент CR1 и удаление одного мономера CentC из массива CentC Mo17. Делеция этого мономера CentC почти наверняка происходила посредством внутрицепочечной репарации DSB на основе гомологии, а не генной конверсии, поскольку последнее потребовало бы почти идеального (по одному повторению) спаривания мономеров CentC между сестринскими хроматидами или ортологичными хромосомами.Если такой механизм спаривания существует в центромерах, было бы удивительно, если бы он не требовал выстраивания вышестоящего элемента xilon (на расстоянии <2 kb) для восстановления точного порядка CentC. Кроме того, тот факт, что точки излома 14 событий неправильного деления центромер риса, сопоставленные с серединой массивов CentO (Cheng et al., 2002), поддерживает частое образование DSB в центромерах.
Мгновенное образование нового мономера CentC путем рекомбинации обеспечивает важный механизм быстрой эволюции тандемных центромерных повторов.Это также предполагает, что преобладание тандемных повторов ДНК в центромерах многих эукариотических хромосом является результатом выбора субстрата, который позволяет эффективно восстанавливать частые DSB в центромерах и около них, вызванные механическим сдвигом, оказываемым на ДНК вблизи веретена микротрубочки (Guerrero et al., 2010). Восстановление DSB было предложено в качестве механизма, который генерировал ряд новых рекомбинантов CR1 у кукурузы (Sharma et al., 2008) и серию новых тандемных повторов около CEN9, которые были получены из CR1 (Sharma et al., 2013), но степень репарации DSB, которая происходит на центромерах, стала очевидной только с доступной сейчас высококачественной последовательностью.
Дивергенция B73 от Mo17 CEN10 на основе этого региона CentC оценивается в 67,9 тыс. Лет назад, что значительно выше, чем предыдущая оценка в 10,3 тыс. Лет назад, полученная из данных HapMap2 (Chia et al., 2012) нерекомбинантного фланкирования. region (Schneider et al., 2016), что повышает вероятность того, что неточная гомология-опосредованная репарация островков CentC может привести к увеличению скорости мутаций в тандемных повторах.Однако, хотя это маловероятно, мы не можем полностью исключить возможность того, что кластеры B73 и Mo17 CentC являются скорее паралогами, чем ортологами.
Альтернативное соединение концов учитывает многие особенности CEN10, включая (1) одиночные CR LTR, (2) внутреннюю делецию CR (рисунок) и делецию мономера CentC (рисунок), (3) добавленную кодирующую последовательность CR (рисунок) и Сегментная дупликация в 22,7 т.п.н. (рисунок) и (4) соседние дупликации, приводящие к CentC HOR (рисунки,). Альтернативная модель соединения концов была предложена с использованием микрогомологически обусловленного соединения концов (MMEJ) в качестве механизма (McVey and Lee, 2008).Полимераза Θ, полимераза, подверженная ошибкам (Arana et al., 2008), была определенно вовлечена в механизм MMEJ (Kent et al., 2015). Эта полимераза подавляет перекрестную рекомбинацию и вызывает остановку клеточного цикла при замалчивании (Ceccaldi et al., 2015). Вполне возможно, что особые механизмы репарации ДНК развились для центромерных областей растений, которые требуют частой репарации.
Гемицентрические инверсии сжимают центромеры и размещают гены в непосредственной близости от них
Мы документируем ряд гемицентрических инверсий, т.е.е., включая один разрыв в активной центромере и второй на плече хромосомы в CEN10. Инверсии наподобие той, которая разделяет начальный кластер CentC на C1 и C2 (Figures,), обладают потенциалом для драматического уменьшения размера активной центромеры и требуют расширения нуклеосом cenh4 на регионы, фланкирующие кластер CentC, чтобы восстановить размер центромеры. По-видимому, именно это произошло в области NC CEN10, которая характеризуется большим количеством недавно вставленных элементов CR.Гемицентрические инверсии относительно коротких фрагментов перетасовывают перицентромерные области, вероятно, с относительно ограниченным эффектом, но те, которые включают длинные области (например, маркеры SyMAP 9 на расстоянии ~ 40 МБ), могут помещать важные гены в активную центромеру (ген 3) и создают потенциал для сильного отбора специфических центромер за счет сцепленного гена. Кроме того, активно транскрибируемые гены связывают меньшее количество cenh4 (Yan et al., 2006) и могут ограничивать экспансию центромер в этом направлении (Wang et al., 2014). Активно транскрибируемый ген 3 длиной> 20 т.п.н. может быть ответственным за ограничение экспансии cenh4 на нижней границе CEN10.
Гемицентрические инверсии были зарегистрированы у кукурузы, в одном случае (обнаруженном FISH) с вовлечением 20% длинного плеча хромосомы 8, что все еще приводило к фертильным гетерозиготам (Lamb et al., 2007b), а в других случаях обнаружено перегруппировкой перицентромерных клеток. маркеры (Wang, Bennetzen, 2012). Наш тщательный анализ CEN10 кукурузы показывает, что гемицентрические инверсии у кукурузы встречаются довольно часто.Используя полученную вручную серию предлагаемых инверсий (рис. S9B), необходимо вызвать не менее 9 проксимальных инверсий по CEN10, поскольку расщепление CentC для учета всех нарушений синтении сорго-кукуруза дает показатель 1 инверсия на <38,9 тыс. Лет назад.
Различные эволюционные силы в центромерах
Наши результаты показывают, что в центромерах эволюция последовательности посредством DSB-индуцированной реаранжировки (делеции, дупликации, инверсии, вставка несинтенических генов или органеллярной ДНК, а также создание рекомбинантных ретротранспозонов и тандемных структур). варианты повторов центромеры) опережает это по единичным нуклеотидным мутациям.По этим и другим причинам (например, храповик Мюллера (Bowers et al., 2005)) центромеры являются плохими соседями для генов. Напротив, гены плохи для центромер, так как они нарушают периодичность тандемных повторов и уменьшают связывание cenh4, если они транскрибируются. Таким образом, разделение хромосом на отдельные бедные генами гетерохроматические перицентрические и богатые генами эухроматические области является логическим следствием этих взаимно антагонистических эффектов. Гемицентрические инверсии потенциально могут нарушить эту хромосомную организацию отдельных территорий.Сходным образом делеция существующей последовательности центромеры с последующим перемещением cenh4 может размещать гены и центромеры в непосредственной близости (Schneider et al., 2016). Наша способность измерять, как гены и центромеры влияют друг на друга и, возможно, влияют на видообразование, улучшится по мере получения дополнительных полных последовательностей центромер для других хромосом и инбредов.
Feizy Rugs Брикстон кукурузный коврик 2′-10 «X 7′-10» Runner Rug | Мебель Sprintz
Коврики keyboard_arrow_right Коврики Feizy Rugs Brixton Maize 2′-10 «X 7′-10» Runner RugАртикул: 6163597FMAZ000I71
Доступно для заказа
Добавить в список Добавить в план комнаты
Ориентировочная цена доставки
Введите свой почтовый индекс, чтобы узнать стоимость доставки.
Представлять на рассмотрение
акция
Копировать ссылку
https://www.sprintz.com/item/brixton-maize-2-10in-x-7-10in-runner-rug/1804747620
Отправить это другу по электронной почте
Бегущий коврик Brixton Maize 2′-10 «X 7′-10» от Feizy Rugs в Sprintz Furniture
Ваше письмо было успешно отправлено.
Закрытое окноПри отправке вашего электронного письма произошла ошибка. Пожалуйста, повторите попытку позже.
Закрытое окно удалитьОписание
Власть вырисовывалась в Турции с ворсом полипропиленового волокна.
Коврик для бега Brixton Maize 2′-10 «X 7′-10», производства Feizy Rugs, представлена вам компанией Sprintz Furniture.Sprintz Furniture — местный мебельный магазин, обслуживающий Нэшвилл, Франклин и район Большого Теннесси. Доступность продукта может отличаться. Свяжитесь с нами, чтобы узнать актуальную информацию о наличии этого продукта. удалитьТехнические характеристики
Доступны дополнительные опции:
| Артикул | Описание | Ссылка |
|---|---|---|
| 6163597FMAZ000A22 | Коврик с кукурузой 2–2 дюйма x 4 фута | Просмотреть товар |
| 6163597FMAZ000I71 | Коврик для бега из кукурузы 2′-10 «X 7′-10» | Текущий товар |
| 6163597FMAZ000h23 | Кукурузный коврик 10 футов X 13 футов 2 дюйма | Просмотреть товар |
| 6163597FMAZ000E10 | Коврик размером 5 x 8 дюймов под кукурузу | Просмотреть товар |
| 6163597FMAZ000G99 | Коврик под кукурузу 8 ‘X 11’ | Просмотреть товар |
Технические характеристики:
| Изделие и размеры | |
|---|---|
| Длина | 94 «L |
| Производитель | Коврики Feizy |
| Длина (фут.) | 7 ’10 « |
| Ширина (фут) | 2 ’10 « |
| Ширина (из стороны в сторону) | 34 дюйма (ширина) |
| Форма | Бегун |
| Информация о конструкции | |
| Материал коврика | Полипропилен |
| Строительная техника | Машинное производство |
| Конструкция Тип | замкнутый |
| Свая | Средний |
| Элементы стиля | |
| Рисунок коврика | геометрический |
| Цвет | золото / желтый |
| Стиль | Переходный |
| Гарантия и информация производителя | |
| Гарантия | 30 дней |
| Происхождение | Импорт |
Похожие поисковые запросы: коврики, коврики для дома, украшения для дома, аксессуары, аксессуары для домашнего дизайна, декоративные коврики, коврики для ванной, большие коврики, маленькие коврики, напольные коврики, уникальные коврики, коврик, домашние коврики, постельный коврик, коврик для гостиной, семья Комнатный Коврик, Кухонные Коврики, Коврик для Прихожей, Акцентный Коврик, Детский Коврик, Комнатные Коврики, Ковер, Декоративные коврики, Напольный коврик, Напольное покрытие, Покрывало, Коврик, Коврик, Бегунок, Ковровые дорожки, Гобелен, Гобелены, Коврик для стола
Номер ссылки: 6163597FMAZ000I71.
Glossy15, APETALA2-подобный ген кукурузы, который регулирует идентичность эпидермальных клеток листа.
- S P Moose and
- P H Sisco
- Департамент генетики Государственного университета Северной Каролины, Роли 27695-7620, США.
Абстракция
Вегетативное развитие многих растений проходит через отдельные ювенильные и взрослые фазы.В кукурузе переход от Развитие побегов от юношеского до взрослого влияет на различные характеристики эпидермальных клеток листа. К ним относятся эпикутикулярные воски, листовые волосы и характеристики клеточной стенки. Предыдущий генетический и фенотипический анализ показал, что ген Glossy15 (Gl15) кукурузы требуется для экспрессии ювенильных эпидермальных признаков после листа 2. Мы сообщаем здесь о молекулярном клонировании Gl15 ген, использующий вставку дефектного элемента супрессора-мутатора (dSpm) в качестве тега транспозона.В соответствии с мутантным фенотипом gl15, характер экспрессии мРНК Gl15 коррелировал с идентичностью эпидермальных клеток ювенильных листьев и регулировался вышестоящим такие факторы, как Corngrass1. Ген Gl15 кодирует предполагаемый фактор транскрипции со значительным сходством последовательности с регуляторные гены Arabidopsis APETALA2 и AINTEGUMENTA, которые действуют в первую очередь для регуляции идентичности органов цветка и семяпочки разработка.Это открытие расширяет известные функции генов, связанных с APETALA2, и включает контроль как вегетативных, так и вегетативных генов. и идентичность репродуктивных боковых органов и обеспечивает молекулярную поддержку гипотезы о том, что листья и цветочные органы являются связанные структуры, полученные из общего плана роста.
Сноски
- Авторские права © Cold Spring Harbor Laboratory Press
Изменчивость микробного сообщества ризосферы при непрерывном производстве семян моно-кукурузы
Заболеваемость на стадии роста и степень тяжести
В целом заболеваемость увеличивалась с увеличением продолжительности посевного года, за исключением 30 лет с стерней (таблица ).Самый высокий уровень заболеваемости фитофторозом всходов и колосовой гнилью составил 8,23% при 20-летнем непрерывном возделывании и 12,92% при 30-летнем непрерывном возделывании без полевой кукурузы, соответственно. Заболеваемость колосовой гнилью при непрерывном земледелии в течение 5 лет была ниже, чем в других случаях, и не наблюдалось разницы между 5 и 10 годами посева. Самый высокий уровень заболеваемости корневой гнилью и гнилью стебля составил 9,58% и 13,55%, соответственно, при непрерывном посеве в течение 20 лет. Очевидно, что непрерывное выращивание семян кукурузы может увеличить заболеваемость с увеличением урожайного года, в то время как заболеваемость четырьмя болезнями различалась в разные годы непрерывного выращивания (Таблица 1).
Таблица 1 Заболеваемость (%) в разные годы непрерывного выращивания сельскохозяйственных культур.Большая часть тяжести заболевания на разных участках разного года сбора урожая характеризовалась уровнем 1 или 2. Видно, что степень тяжести заболевания — фитофтороз, стеблевая гниль и колосовая гниль, которые были обычным заболеванием в районе кукурузы. урожай семян был высоким в большинство посевных лет. Серьезность заболевания колосовой гнилью увеличивалась с годом сбора урожая, в то время как серьезность заболевания снижалась к 30 годам из-за фитофтороза всходов и корневой гнили кукурузы на полях.Тяжесть корневой гнили была относительно небольшой по сравнению с другими болезнями почти во все разные годы выращивания сельскохозяйственных культур (Таблица 2).
Таблица 2 Степень тяжести различных заболеваний в разные годы непрерывного земледелия.Разнообразие сообществ грибов после выращивания семян кукурузы на моно последовательности.Для представления реального разнообразия микробов в образцах были выбраны Чао 1, наблюдаемые виды, PD-целое дерево и индекс Шеннона (рис. 1).
Рис. 1Chao1, наблюдаемые виды, PD_whole_tree и индекс Шеннона грибов разных непрерывных сельскохозяйственных культур. Заглавные буквы A, B, C, D, E, F и G означают 5, 10, 15, 20, 25, 30 и 30 лет с стерни соответственно.
Индексы Chao1, PD-целое дерево, индекс Шеннона имели 97% -ное сходство.Самый высокий и самый низкий индекс Шеннона, наблюдаемые виды, Chao1, наблюдались через 30 и 10 лет возделывания почвы соответственно (рис. 1). Количество наблюдаемых OTU увеличивалось с увеличением количества выбранных последовательностей и достигло области близкого к плато при уровне сходства 97%. Кривые разрежения имели тенденцию к непрерывному увеличению по мере увеличения количества последовательностей (рис. 2A), за исключением 172. общих OTU, верхний предел OTU для 5, 10, 15, 20, 25 и 30 сельскохозяйственных лет составлял 703, 619, 630, 785, 976, 1056 и 922 соответственно (рис.2Б). Все кривые ранг-численность уменьшены и сглажены в конце, а по оси абсцисс находятся более 10 лет, показывая более высокую численность через 30 и 25 лет, с максимальным значением за 30 лет (рис. 2С). Непрерывное земледелие обогатило разнообразие и богатство грибного сообщества, и разнообразие увеличивалось за годы непрерывного земледелия, за исключением 10-летнего земледелия.
Рисунок 2( A ) Кривые разрежения OTU, сгруппированные с идентичностью последовательностей <97% для 21 различных образцов почвы с ризошпером для производства семян кукурузы за 5, 10, 15, 20, 25, 30 и 30 лет непрерывного возделывания .Заглавные буквы A, B, C, D, E, F и G означают 5, 10, 15, 20, 25, 30 и 30 лет соответственно. Нижний индекс под буквой представляет три дубликата одного и того же урожайного года. Показания OTU различных образцов показаны разными цветами. ( B ) Цветочный график OTU разного непрерывного сельскохозяйственного года. Прописные буквы A, B, C, D, E, F и G обозначают 5, 10, 15, 20, 25, 30, 30 лет соответственно. Ядром были общие ОТЕ для разного лечения. ( C ) Кривые относительного обилия OTU для OTU в 21 различных образцах почвы для производства семян кукурузы с ризошпером за 5, 10, 15, 20, 25, 30 и 30 (с полевой кукурузой) лет непрерывного возделывания.Прописные буквы A, B, C, D, E, F и G обозначают 5, 10, 15, 20, 25, 30 и 30 лет соответственно. Нижний индекс под буквой представляет три дубликата одного и того же урожайного года. Показания OTU различных образцов показаны разными цветами.
Относительная численность грибного сообщества
Разнообразие и относительная численность грибного сообщества ризосферы значительно различались в зависимости от года непрерывного сбора урожая. На уровне филума преобладали последовательности грибов, распространенные у Ascomycota, Mortierellomycota, Basidiomycota, Glomeromycota и Rozellomycota.В частности, Ascomycota была наиболее доминирующим филумом грибов и составляла в среднем 65,98% всей последовательности ДНК грибов. На пять известных типов грибов в среднем приходилось 76,09–89,01% от всех. По-прежнему присутствует 12,45–23,91% неидентифицированных и других грибковых типов, а количество неидентифицированных типов увеличивалось с увеличением продолжительности года выращивания урожая (рис. 3A). На уровне рода количество результатов сортировки, упорядоченных от высокого к низкому, составляло Mortierella, Chrysosporium, Pseudeurotium, Fusarium, Podospora, Chaetomium, Exophiala, Bipolaris, Gibberella, Acremonium, Clasosporium, Microascus, Cladorhomyces, G Plectosphaerella, Wardomyces, Tetracladium, Inocybe, Holtermanniella, и Bionectria , принадлежащие к разным типам.Значительные виды грибов на ризосферной почве не были классифицированы и показали относительный рост за 20 лет посевов. Определенная доля потенциальных патогенных микроорганизмов Fusarium, Gibberella, Cladosporium, Acremonium, и Bipolaris была обнаружена в ризосферной почве разного непрерывного сельскохозяйственного года. Стоит отметить, что относительная численность Fusarium, Gibberella, Cladosporium, Exophiala и относительное истощение Mortierella, Podospora, Chrysosporium и Chaetomium в разные годы непрерывной посадки урожая (рис.3Б). Согласно анализу Лефсе, вид Gibberella-Baccata был обогащен ризосферной почвой с 30-летним урожаем и 3-летней историей посева кукурузы в поле. Ключевой род Fusarium, Curvularia, Currlaria-lunata доминировал в течение 30 лет непрерывного земледелия, а род Chaetochyriales доминировал в течение 25 лет. Виды Mortierella-Elongata в почве 20-летнего возделывания и род plectosphaerellaceae в почве 15-летнего непрерывного возделывания были более обогащенными, чем другие виды обработки.Класс Mortierellamycetes и виды Mortierella_sp были чрезмерно представлены в ризосферной почве с 10- и 5-летним непрерывным сельскохозяйственным годом, и эти ключевые типы потенциально способствовали структурной сегрегации в разные годы (рис. 3C).
Рис. 3( A ) Состав различных грибных сообществ на уровне филума в семи образцах почвы ризосферы. Относительное обилие различных типов грибов в разных сообществах показано разными цветами.Неидентифицированный означает отсутствие таксономической информации, а Другой означает, что группа с множеством функций. ( B ) Состав различных грибных сообществ на уровне рода в семи образцах почвы ризосферы. Относительная численность разных видов грибов в разных сообществах показана разными цветами. Неидентифицированный означает отсутствие таксономической информации, а Другой означает, что группа с множеством функций. ( C ) Оценка LDA грибов разного года сбора урожая. На рисунке показаны виды, у которых оценка LDA больше установленного значения (оценка LDA> 2, менее строгий установлен на 2; более строгий установлен на 4), то есть биомаркеры со статистическими различиями между группами.Длина столбчатой диаграммы отражает значимость различных видов.
Разнообразие сообществ бактерий после монопрерывного выращивания
Результаты отражают значительные различия между образцами в составе сообщества бактерий ( P <0,05), а также более высокий индекс Чао1 и Шеннона за 30 лет и 10 лет урожая с самым высоким Наблюдались 30-летние посевы без полевой кукурузы. Двадцатипятилетний урожай показал самый высокий уровень, в то время как пятилетний урожай показал самые низкие наблюдаемые виды и дерево PD (рис.4А). Восемнадцать тысяч оптимизированных последовательностей со средней длиной 200–260 п.н. были сохранены путем фильтрации из 21 000 действительных последовательностей. Кривые разрежения имели тенденцию к уменьшению по мере увеличения количества последовательностей, а верхний предел OTUs 0,03 составлял 2735, 2952, 2415, 2655, 2679, 3220 и 2805 соответственно. Диаграмма Венна показала, что среди непрерывно обрабатываемых образцов почвы было 743 общих ОТЕ, а через 5, 10, 15, 20, 25, 30 и 30 лет было обнаружено 2296, 2535, 2543, 2426, 2451, 2645 и 2378 других ОТЕ. сплошная обрезка (рис.4Б). Как показало высокопроизводительное секвенирование, непрерывное земледелие вызвало разное количество ОТЕ, а бактериальное разнообразие и структура сообщества варьировались в разные годы непрерывного возделывания.
Рис. 4( A ) Chao1, наблюдаемые виды, PD_ целое дерево и индекс Шеннона бактерий разных непрерывных сельскохозяйственных культур. Заглавные буквы A, B, C, D, E, F и G соответствуют 5, 10, 15, 20, 25, 30 и 30 годам обрезки соответственно. ( B ) Диаграмма Венна бактерий разного непрерывного урожая года.A, B, C, D, E, F и G соответствуют 5, 10, 15, 20, 25, 30 и 30 годам сбора урожая соответственно.
Относительное обилие бактерий ризосферной почвы
Относительное обилие заданных типов и родов бактерий сравнивалось в различных образцах (рис. 5). Результаты показали, что Proteobacteria, Actinobacteria, Chloroflexi, Planctomycetes и Acidobacteria находятся в первой пятерке среди всех типов бактерий, которые составляют 22,46–26,71%, 15,70–24,43%, 11,6–15.45%, 8,65–10,91% и 8,07–10,38% разного урожая, соответственно. Proteobacteria обогащались ризосферными почвами при 10-летнем возделывании, тогда как Gemmatimonadetes и Thaumarchaeota истощались при 10-летнем возделывании. Протеобактерии — ключевой тип ризосферной почвы при непрерывном производстве семян кукурузы (рис. 5А). Два рода Flavobacterrium и Leptolyngbya показали более низкую численность, в то время как Pseudarthrobacter, RB41 и Roseiflexus показали более высокую численность в различных образцах почвы, относительная численность которых составляла 0.02–0,72%, 0–1,32%, 0,97–5,75%, 0,77–2,43% и 0,71–2,23% соответственно. Haliangium и Streptomyces также присутствовали в почвах, которые занимали 0,84–1,4% и 0,38–0,86% в разные сельскохозяйственные годы (рис. 5B). Многолетний посев семян кукурузы увеличил количество Roseiflexus и Blastococcus в почве ризосферы кукурузы. Непрерывное земледелие привело к большим изменениям бактериальной флоры в почве ризосферы кукурузы. Согласно анализу Lefse, актинобактерии и цианобактерии преобладали при выращивании в течение 5–10 лет, в то время как Chloroflexi и Roseiflexus были чрезмерно представлены при выращивании в течение 20 лет. Blastocatellaceae-subgroup-4 и Rhodocyclaceae доминировали при выращивании урожая 25 лет, Thermoleophilia и Solirubrobacterales преобладали при 30-летнем возделывании кукурузы, в то время как Chloroflexi и Bacilli преобладали при возделывании кукурузы 30 лет (Рис. 5C). Для более глубокого понимания различий между группами был применен сетевой анализ профильной кластеризации. Профиль, полученный с помощью сетевого анализа Cytoscape, показал все типы и выделил распределение и относительную численность всех типов в разные годы выращивания (рис.6А, Б). Актинобактерии были обогащены в течение пяти лет, в течение 10 лет — с относительным обилием Firmicutes, в течение 15 лет — с повышенным содержанием нитроспир и тектомикробов, а в течение 30 лет — с выделением Chloroflexi. На уровне семьи, 10 лет выращивания обогащали Geodermatophilaceae и OM1_clade, 15 лет выращивания обогащали Blastocatellia и OM1_clade. 30 лет выращивания полевой кукурузы, обогащенной Blastocatellaceae_sugbgoup_4, Geodermatophilaceae, Blastocatellia, OM1_clade, Geodermatophilaceae, Frankiales, Pseudonocardiaceae и Cellulomatophilaceae, в то время как 30 лет выращивания без полевой кукурузы, Frankiales, обогащенной Blastocatelliales.Отчетливо наблюдалась значительная разница в относительной численности в разные годы сбора урожая у разных филумов и родов ( P <0,01). OTU_20 Proteobacteria больше коррелировал с OTU_6 Chloroflexi, а OTU_2 Thaumarchaeota больше коррелировал с OTU-45 Actinobacteria. Также было ясно показано совместное появление разных типов (рис. 6А, В).
Рис. 5( A ) Бактериальный тип разных лет выращивания на основе среднего числа считываний на образец.( B ) Бактериальный род разных лет выращивания на основе среднего числа считываний на образец. ( C ) Оценка LDA бактерий разного года сбора урожая. На рисунке показаны виды, у которых оценка LDA больше установленного значения (оценка LDA> 2, менее строгая установлена на 2; более строгая была установлена на 4), то есть биомаркеры со статистическими различиями между группами. Длина столбчатой диаграммы отражает значимость различных видов.
Рисунок 6( A ) Кладограмма почвенных ризосферных бактерий, генерируемых LEfSe, показывающая различия таксонов между обработками.Связь означает сильное и значительное событие. Размер каждого узла пропорционален относительной численности. Красный, темно-зеленый, синий, пурпурный, зеленый, оранжевый и светло-зеленый цвета на бактериальных деревьях указывают на таксоны, которые обогащены полевой кукурузой в год сбора урожая 5, 10, 15, 20, 25, 30 и 30 соответственно ( ɑ> 0,01). ( B ) Совместное сетевое взаимодействие почвенных ризосферных бактерий. Связь означает сильную и значительную взаимосвязь.Размер каждого узла пропорционален относительной численности.
Взаимосвязь между заболеваемостью и составом микробного сообщества
По результатам RDA грибков, корневая гниль, фитофтороз и початковая гниль показали аналогичные тенденции изменения, и их заболеваемость была значимо положительно связана с Podospora, Trichoderma, Tetracladium и Exophiala . В то время как вышеупомянутые заболевания были значительно отрицательно связаны с Microascus, Cladosporium, Acremonium, Chaetomium, и Pseudeurotium .Стеблевая гниль была значимо положительно связана с Fusarium, Gibberella, Bipolaris, Plectosphaerella , в то время как заболеваемость ею была значимо отрицательно коррелирована с Wardomyces, Chrysosporium, Curvularia и Holtermanniella . Различные патогенные патогены приводили к разной заболеваемости в разные годы непрерывного выращивания (рис. 7A).
Рисунок 7( A ) RDA-анализ взаимосвязи между заболеваемостью и относительной численностью грибов разных лет выращивания.Корреляция между различной заболеваемостью и осями RDA была представлена длиной и углом стрелок. ( B ) RDA-анализ взаимосвязи между заболеваемостью в разные годы непрерывного выращивания и относительной численностью бактерий разных лет. Корреляция между различной заболеваемостью и осями RDA была представлена длиной и углом стрелок.
По результатам RDA по бактериям, частота заболевания при непрерывном выращивании в течение 5 лет была достоверно положительно коррелирована с Pseudarthrobacter, Gemmata, Steroidobacter, Pirellula, Sphingomonas, Halianggium, Nocardioides, Clostridiu, Pir4-lineage, Pirecesllula, 20 Streptomy и 10-летний урожай был положительно связан со всеми вышеупомянутыми бактериями.Непрерывное выращивание в течение 15 лет было достоверно положительно связано с Flavobacterrium и Leptolyngbya , а 20-летнее выращивание было положительно связано с Pseudarthrobacte . 30-летний урожай был положительно связан с Planctomyces, Flavobacterrium и родом U не идентифицирован , а 30-летний урожай имел значительную положительную связь с ними. Непрерывное выращивание кукурузы в течение 25 и 30 лет было достоверно положительно связано с родами Roseiflexus, Blastococcus, Solirubrobacter, RB41, и Skermanella (рис.7Б).
Новый подход к генетической модификации кукурузы повышает урожайность на 10 процентов
Урожайность кукурузы (кукурузы) можно увеличить на целых 10 процентов, изменив один ген, чтобы он включился раньше, чем это обычно происходит в цикле роста растения, новое исследование заключает.
Результаты важны, потому что кукуруза является наиболее широко выращиваемой зерновой культурой в мире, служащей основным продуктом питания для миллионов людей, а также домашнего скота. Он также имеет множество промышленных применений. Но ожидается, что текущая доходность не будет соответствовать спросу.
«В ближайшие десятилетия продовольственная безопасность, вероятно, станет более серьезной проблемой, поскольку будет расти спрос на зерно, который необходимо будет удовлетворять экологически устойчивым образом», — написали исследователи в своем резюме. «На сегодняшний день коммерческая трансгенная кукуруза имеет прежде всего направленную устойчивость к насекомым и гербицидам. Здесь мы описываем трансгенный подход к повышению урожайности и стабильности урожая кукурузы ».
Исследование, опубликованное в PNAS, было сосредоточено на zmm28 , гене, который включается, когда растение кукурузы начинает цвести.Используя промоутер, исследователи добились более раннего включения zmm28 , что, по сути, заставило его работать усерднее и дольше.
Исследователи протестировали усиленный ген на 48 различных гибридах кукурузы в течение трех лет в различных условиях выращивания в Соединенных Штатах. Урожайность растений обычно была на 3-5 процентов выше, чем у контрольных групп, при этом некоторые генетически модифицированные гибриды давали на 8-10 процентов больше зерна.
Повышение урожайности связано с несколькими факторами.Сконструированные растения вырастили немного более крупные листья, которые лучше справляются с фотосинтезом, а также они были на 16-18 процентов эффективнее в использовании азота.
«[Эти] трансгенные растения продемонстрировали, что их улучшенные агрономические характеристики связаны с повышенной ассимиляцией углерода растениями, использованием азота и ростом растений», — говорится в аннотации. «В целом, эти положительные характеристики связаны со значительным увеличением урожайности зерна по сравнению с контролями дикого типа, которое остается неизменным по годам, окружающей среде и элитному фону зародышевой плазмы.”
Исследователи надеются, что их работа может быть применена к другим зерновым культурам. Corteva уже добивается одобрения новых сортов кукурузы в Министерстве сельского хозяйства США и оценивает, что для управления процессами регулирования по всему миру может потребоваться от шести до 10 лет.
Категории.

 0 (Marshmallow)
Два четырехъядерных процессора с частотой 1,5 и 1 ГГц
0 (Marshmallow)
Два четырехъядерных процессора с частотой 1,5 и 1 ГГц процессор
процессор процессор
процессор 0
0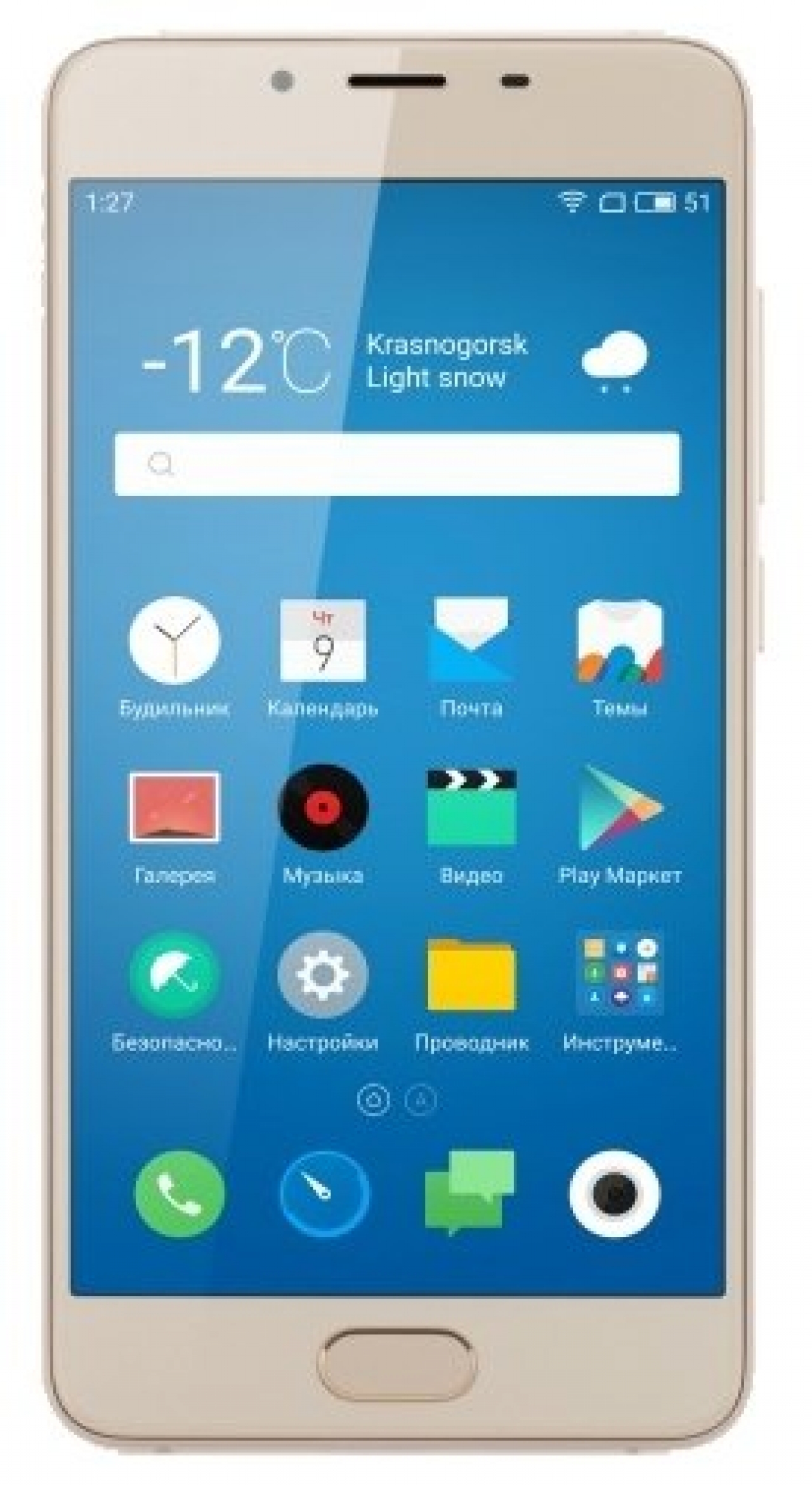 процессор
процессор 0
0 0
0 0
0 0
0 0
0 0
0 1
1 0
0




 99 грн.
99 грн.










 У него недавний и нестабильный геном с необычайной изменчивостью в популяции. Поле содержит семьи, несущие ~ 100 различных генетических мутаций, каждая из которых может вызывать различный, интересный, а иногда и странный фенотип (внешний вид). Многие из этих естественных мутантов были обнаружены более 50 лет назад.Это похоже на ботанический карнавал уродов … Шаг вперед! Посмотрите на 6-дюймовых карликов , полосатых растений , имитаторов поражений , растений-призраков , знаменитых узловатых мутантов и gnarley , а также любимых лабораторий, ленивых мутантов , которые предпочли бы лягте на землю, чем встаньте.
У него недавний и нестабильный геном с необычайной изменчивостью в популяции. Поле содержит семьи, несущие ~ 100 различных генетических мутаций, каждая из которых может вызывать различный, интересный, а иногда и странный фенотип (внешний вид). Многие из этих естественных мутантов были обнаружены более 50 лет назад.Это похоже на ботанический карнавал уродов … Шаг вперед! Посмотрите на 6-дюймовых карликов , полосатых растений , имитаторов поражений , растений-призраков , знаменитых узловатых мутантов и gnarley , а также любимых лабораторий, ленивых мутантов , которые предпочли бы лягте на землю, чем встаньте. Приходите прогуляться по мутантам кукурузы. Предоставляется бесплатный обед и холодные напитки.
Приходите прогуляться по мутантам кукурузы. Предоставляется бесплатный обед и холодные напитки. Д., Сделанные:
Д., Сделанные:  которые изначально были выделены генетиками, селекционерами и фермерами
за последние 80 лет.Многие из них считаются классическими мутантами и изучаются годами.
узнать о принципах и механизмах генетики и наследственности.
Запасы мутантов распространяются среди ученых и селекционеров через Каталог фондового центра сотрудничества в области генетики кукурузы.
Почти все запасы кукурузы были получены HWB для
Проект «Кукуруза-10-Лабиринт» из цеха по выращиванию кукурузы зимой 2003/04 г.
которые изначально были выделены генетиками, селекционерами и фермерами
за последние 80 лет.Многие из них считаются классическими мутантами и изучаются годами.
узнать о принципах и механизмах генетики и наследственности.
Запасы мутантов распространяются среди ученых и селекционеров через Каталог фондового центра сотрудничества в области генетики кукурузы.
Почти все запасы кукурузы были получены HWB для
Проект «Кукуруза-10-Лабиринт» из цеха по выращиванию кукурузы зимой 2003/04 г. com
com